接種時期選定の参考 (新)


このページには、覚えないといけないことは特に何もありません。
3-3-1.武漢株(Wuhan-Hu-1)をチラ見する
3-3-2.オミクロンの出現
3-3-3.令和5年2023年1月頃の変異
3-3-4.令和6年2024年1月頃の変異
次図は細胞内における「DNAの遺伝情報」→「RNAの遺伝情報」→「タンパク合成(アミノ酸配列の情報)」の略図です。
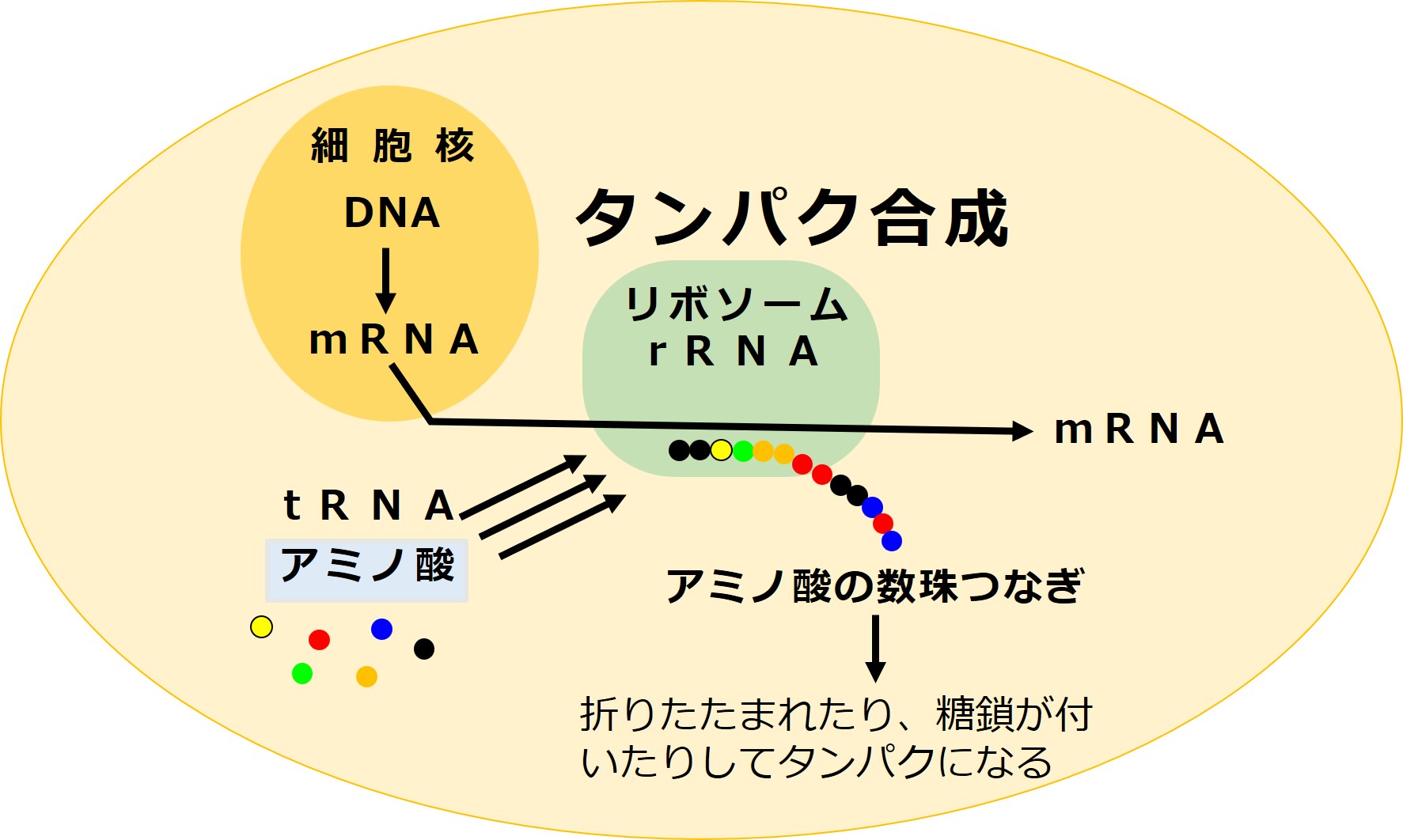
ここで使われるDNAの塩基は4種類(AGTC)、RNAの塩基も4種類(AGUC)あります。
タンパク合成に使われるアミノ酸は20種類(ACDEFGHIKLMNPQRSTVWY)あります。
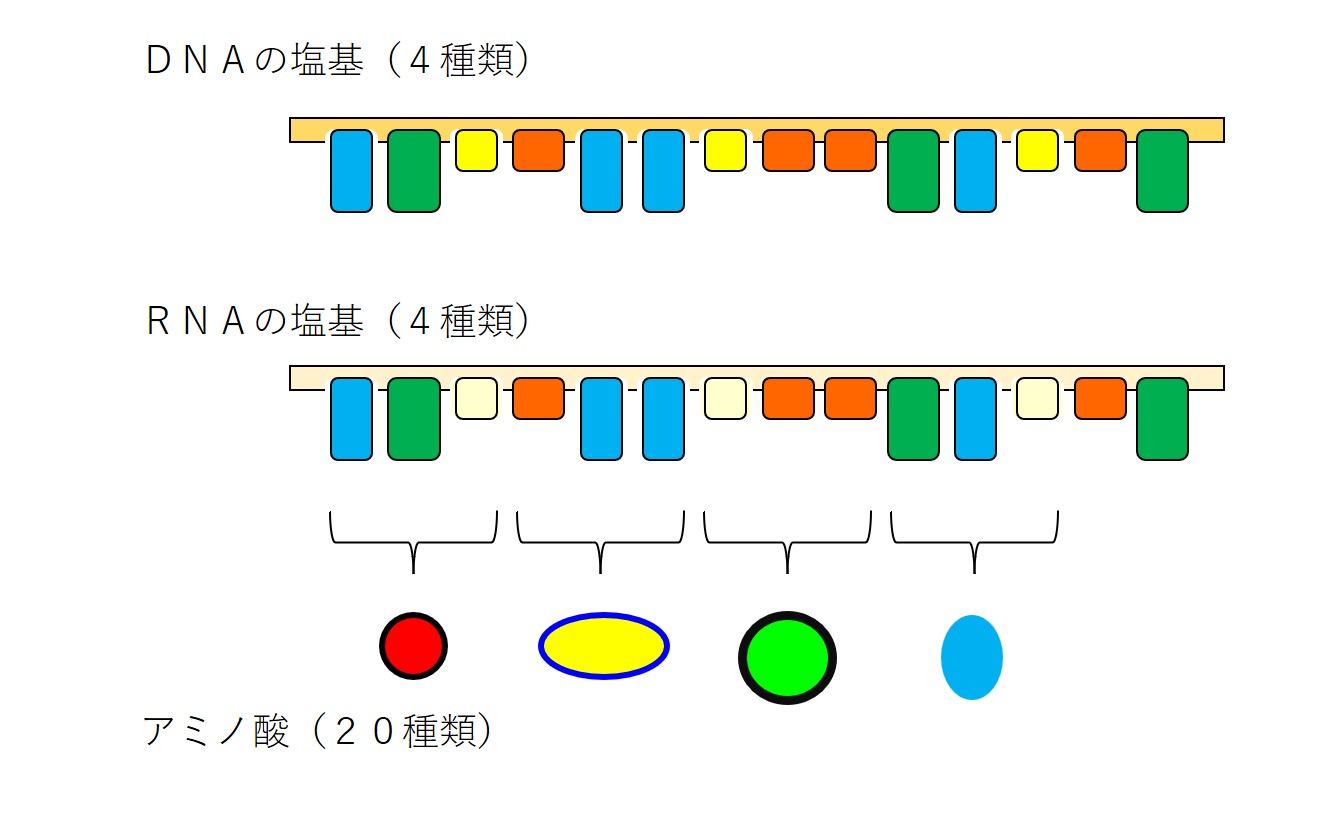
「塩基配列3個の1組」と「アミノ酸1個」が対応しています。この対応表を コドン表 と呼びます。
では、まずは武漢株(Wuhan-Hu-1, 2020.07.18報告版)の塩基配列やアミノ酸配列を見てみましょう。
右をクリックすると下図のページに移動しますが:▶ ▶ ▶(Severe acute respiratory syndrome coronavirus 2 isolate Wuhan-Hu-1, complete genome)
先に、ページの案内を見てください:↓
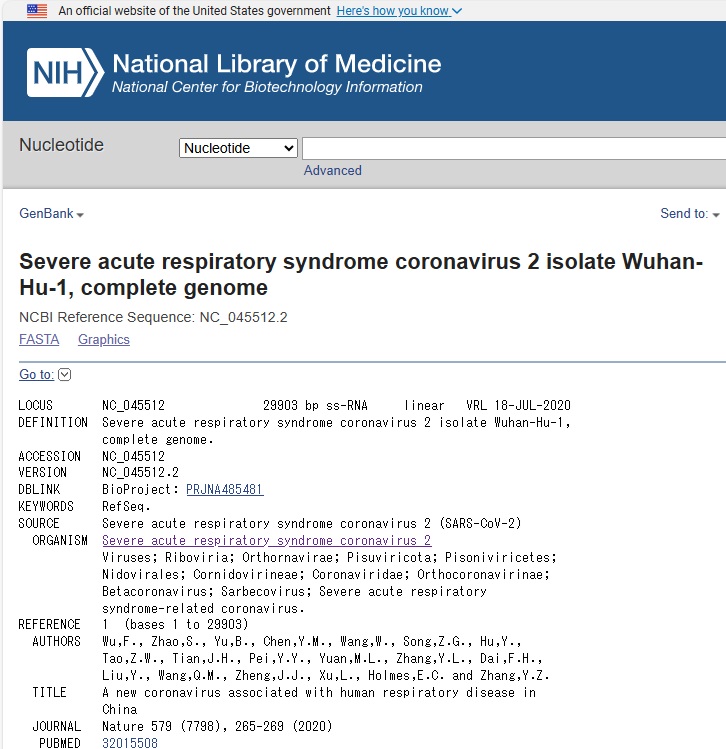
塩基の総数は「29903」個であり「1番から29903番まで」番号が付与されています。
ページの下の方(ページの半分位の位置)に移ると「/gene="S"」と書かれた部分があります。
そこにコロナのスパイク(Sタンパク)の配列情報があります。
塩基の番号「21563番~25384番」までがスパイクのアミノ酸配列の情報を持っています。
「/translation="MFVFLVLLPLVSSQ……」のところにアミノ酸配列情報があります。スパイクのアミノ酸は「MFVFLV……」となっています。
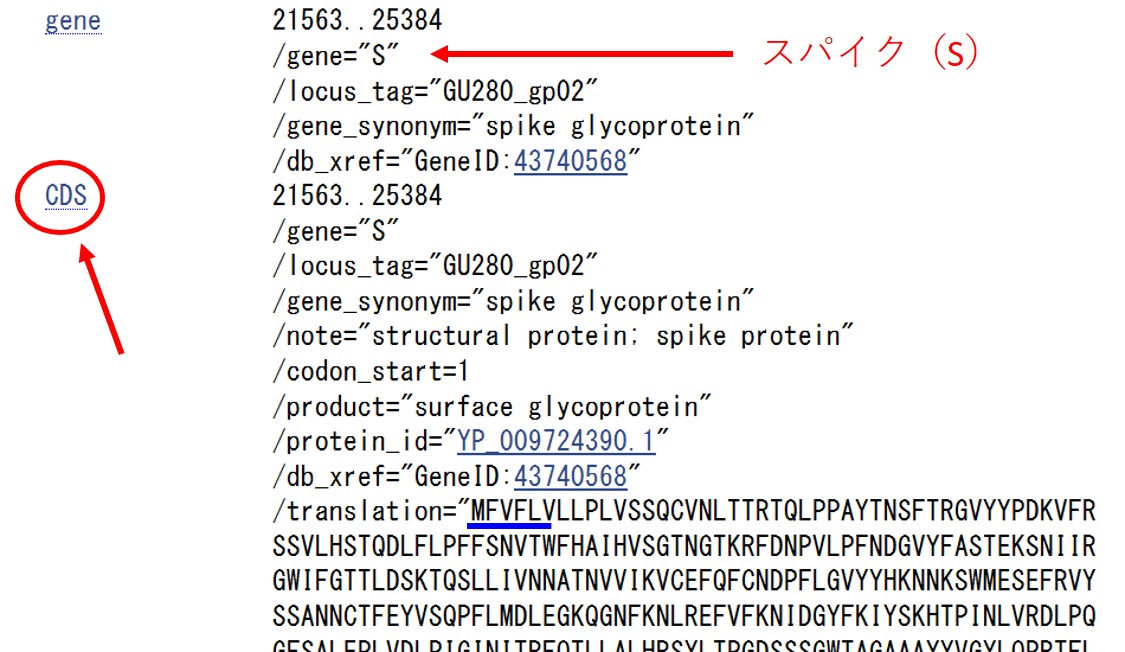
左側にある「CDS」をクリックすると、ページ下方にある塩基配列の中でスパイクをコードする塩基が強調表示されます。コロナはRNAウイルスですが、塩基はDNAの塩基(AGTC)で記録されています。
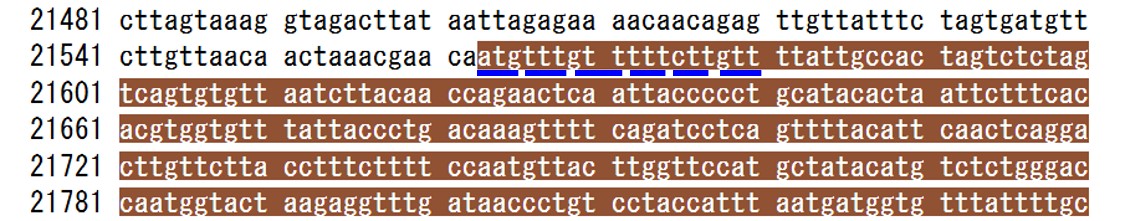
コドン表を使って塩基配列とアミノ酸配列を見比べると、きちんと対応していることがわかります。

では、実際に武漢株(2020.07.18報告版)のページを見てみましょう:▶ ▶ ▶(Severe acute respiratory syndrome coronavirus 2 isolate Wuhan-Hu-1, complete genome):この武漢株(2020.07.18報告版)が新型コロナウイルス SARS-CoV-2 の変異を見る基準になっているようです。
こちらは武漢株(2020.01.13報告版)です:▶ ▶ ▶(Wuhan seafood market pneumonia virus isolate Wuhan-Hu-1, complete genome)
また、横浜に来たダイアモンド・プリンセス号の感染者から得たウイルスの配列情報も見てみましょう:▶ ▶ ▶ (神奈川県衛生研究所)
次の表は、武漢株(2020.07.18報告版)のスパイクのアミノ酸配列を 40 個ごとに区切って表にしたものです。
10 個毎に半角スペースを入れてあります。
RBD(受容体結合部位)を構成する 319 番 ~ 541 番のアミノ酸を赤字にしています。
「614番目のアミノ酸:D」には黄色いマークを付けています。
| No. | アミノ酸配列 | 個数 |
| 1 | MFVFLVLLPL VSSQCVNLTT RTQLPPAYTN SFTRGVYYPD | 40 |
| 41 | KVFRSSVLHS TQDLFLPFFS NVTWFHAIHV SGTNGTKRFD | 40 |
| 81 | NPVLPFNDGV YFASTEKSNI IRGWIFGTTL DSKTQSLLIV | 40 |
| 121 | NNATNVVIKV CEFQFCNDPF LGVYYHKNNK SWMESEFRVY | 40 |
| 161 | SSANNCTFEY VSQPFLMDLE GKQGNFKNLR EFVFKNIDGY | 40 |
| 201 | FKIYSKHTPI NLVRDLPQGF SALEPLVDLP IGINITRFQT | 40 |
| 241 | LLALHRSYLT PGDSSSGWTA GAAAYYVGYL QPRTFLLKYN | 40 |
| 281 | ENGTITDAVD CALDPLSETK CTLKSFTVEK GIYQTSNFRV | 40 |
| 321 | QPTESIVRFP NITNLCPFGE VFNATRFASV YAWNRKRISN | 40 |
| 361 | CVADYSVLYN SASFSTFKCY GVSPTKLNDL CFTNVYADSF | 40 |
| 401 | VIRGDEVRQI APGQTGKIAD YNYKLPDDFT GCVIAWNSNN | 40 |
| 441 | LDSKVGGNYN YLYRLFRKSN LKPFERDIST EIYQAGSTPC | 40 |
| 481 | NGVEGFNCYF PLQSYGFQPT NGVGYQPYRV VVLSFELLHA | 40 |
| 521 | PATVCGPKKS TNLVKNKCVN FNFNGLTGTG VLTESNKKFL | 40 |
| 561 | PFQQFGRDIA DTTDAVRDPQ TLEILDITPC SFGGVSVITP | 40 |
| 601 | GTNTSNQVAV LYQDVNCTEV PVAIHADQLT PTWRVYSTGS | 40 |
| 641 | NVFQTRAGCL IGAEHVNNSY ECDIPIGAGI CASYQTQTNS | 40 |
| 681 | PRRARSVASQ SIIAYTMSLG AENSVAYSNN SIAIPTNFTI | 40 |
| 721 | SVTTEILPVS MTKTSVDCTM YICGDSTECS NLLLQYGSFC | 40 |
| 761 | TQLNRALTGI AVEQDKNTQE VFAQVKQIYK TPPIKDFGGF | 40 |
| 801 | NFSQILPDPS KPSKRSFIED LLFNKVTLAD AGFIKQYGDC | 40 |
| 841 | LGDIAARDLI CAQKFNGLTV LPPLLTDEMI AQYTSALLAG | 40 |
| 881 | TITSGWTFGA GAALQIPFAM QMAYRFNGIG VTQNVLYENQ | 40 |
| 921 | KLIANQFNSA IGKIQDSLSS TASALGKLQD VVNQNAQALN | 40 |
| 961 | TLVKQLSSNF GAISSVLNDI LSRLDKVEAE VQIDRLITGR | 40 |
| 1001 | LQSLQTYVTQ QLIRAAEIRA SANLAATKMS ECVLGQSKRV | 40 |
| 1041 | DFCGKGYHLM SFPQSAPHGV VFLHVTYVPA QEKNFTTAPA | 40 |
| 1081 | ICHDGKAHFP REGVFVSNGT HWFVTQRNFY EPQIITTDNT | 40 |
| 1121 | FVSGNCDVVI GIVNNTVYDP LQPELDSFKE ELDKYFKNHT | 40 |
| 1161 | SPDVDLGDIS GINASVVNIQ KEIDRLNEVA KNLNESLIDL | 40 |
| 1201 | QELGKYEQYI KWPWYIWLGF IAGLIAIVMV TIMLCCMTSC | 40 |
| 1241 | CSCLKGCCSC GSCCKFDEDD SEPVLKGVKL HYT | 33 |
| 1273 |
ウイルスの変異によってタンパクのアミノ酸配列が変化したときは次のように表記します(例):D614G

詳しく見たい人向けの参考
コロナウイルスの変異を理解する Nature ダイジェスト Vol.17 No.12 2020年の記事です。
新型コロナウイルス(SARS-CoV-2)の変異 増田道明 2021年の記事です。
まず、オミクロン株登場の当初、世界がその不自然な変異ぶりに驚いた様子を伝える記事を紹介しておきます(注 → 記事をじっくり読む必要はありません。サーっと眺めて、みんなビックリしたんだなあ、とわかればそれで構いません):
「まずい変異がてんこ盛り オミクロン型出現のわけ」 (日本経済新聞、2021.12.24、12月25日発売の日経サイエンス2022年2月号、出村政彬)
「オミクロン型出現 変異箇所の多さから浮かぶ3つの仮説」 (日本経済新聞、2022.01.14、日経サイエンス2022年2月号、出村政彬)
日経サイエンスの図↓↓↓
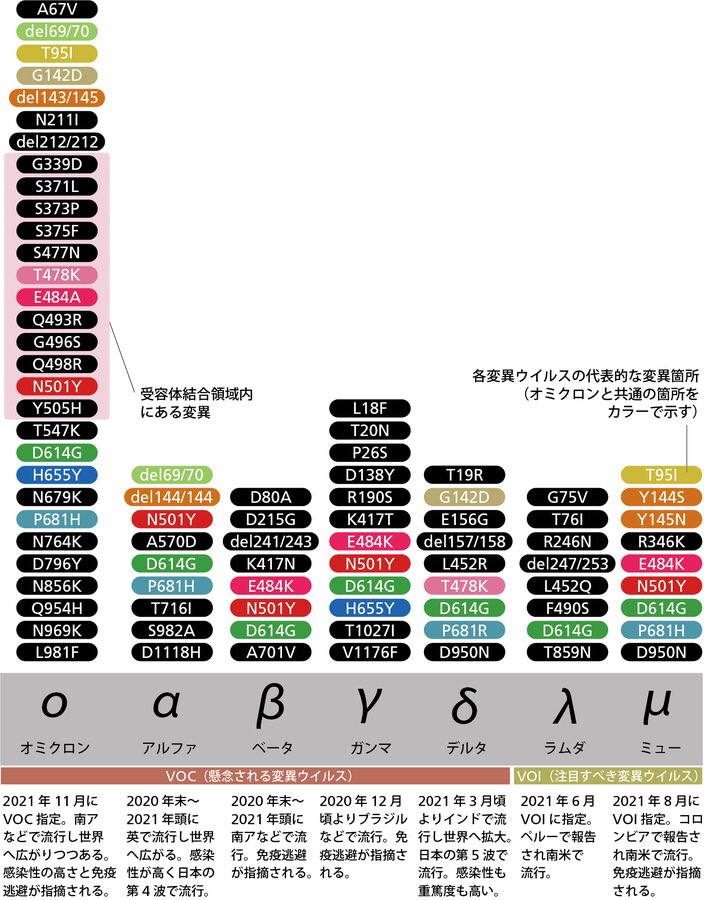
この謎を鮮やかに解いたのが 荒川央(あらかわひろし)さんです。
高等学校の生物基礎程度の知識があり、「DNA→RNA→タンパク」の流れについてご存じの方は、そのまま荒川央(あらかわひろし)さんの記事を読んでください。難なく理解できると思われます。
荒川央さんは免疫学者です。荒川央さんによる分析の圧巻は「オミクロン変異考察」です。コロナウイルス(オミクロン株)が天然のウイルスではない証拠が示されています。
荒川央さんのブログ(2021.06.08~2022.01.17)は書籍化されています。(高校の生物基礎+アルファの知識が必要) 「コロナワクチンが危険な理由」、荒川央、花伝社(税抜き¥1,500)、2022.03.25
プレプリント Mutation signature of SARS-CoV-2 variants raises questions to their natural origins. Arakawa, Hiroshi June 1, 2022
ここからは、「DNA→RNA→タンパク」についてあまり知らない方に向けて、「オミクロン変異考察」の要点を理解できるように基本的なことから説明します。
ほんの少しのことを理解するだけで、荒川央さんの記事を読めるようになりますので、以下はサーっと読み通していってください。
まず、全体のイメージを把握していただくために、ざくっと概要を紹介します。なんとなく雰囲気が分かればよいので、知らない用語があっても無視して読み飛ばしましょう。
コロナウイルスはRNAウイルスです。RNAの 塩基配列 でウイルスタンパクの アミノ酸配列 が決まります。
RNAの塩基配列では、時々 突然変異 が起こって塩基が入れ替わっています。この変異は自然に起こっています。
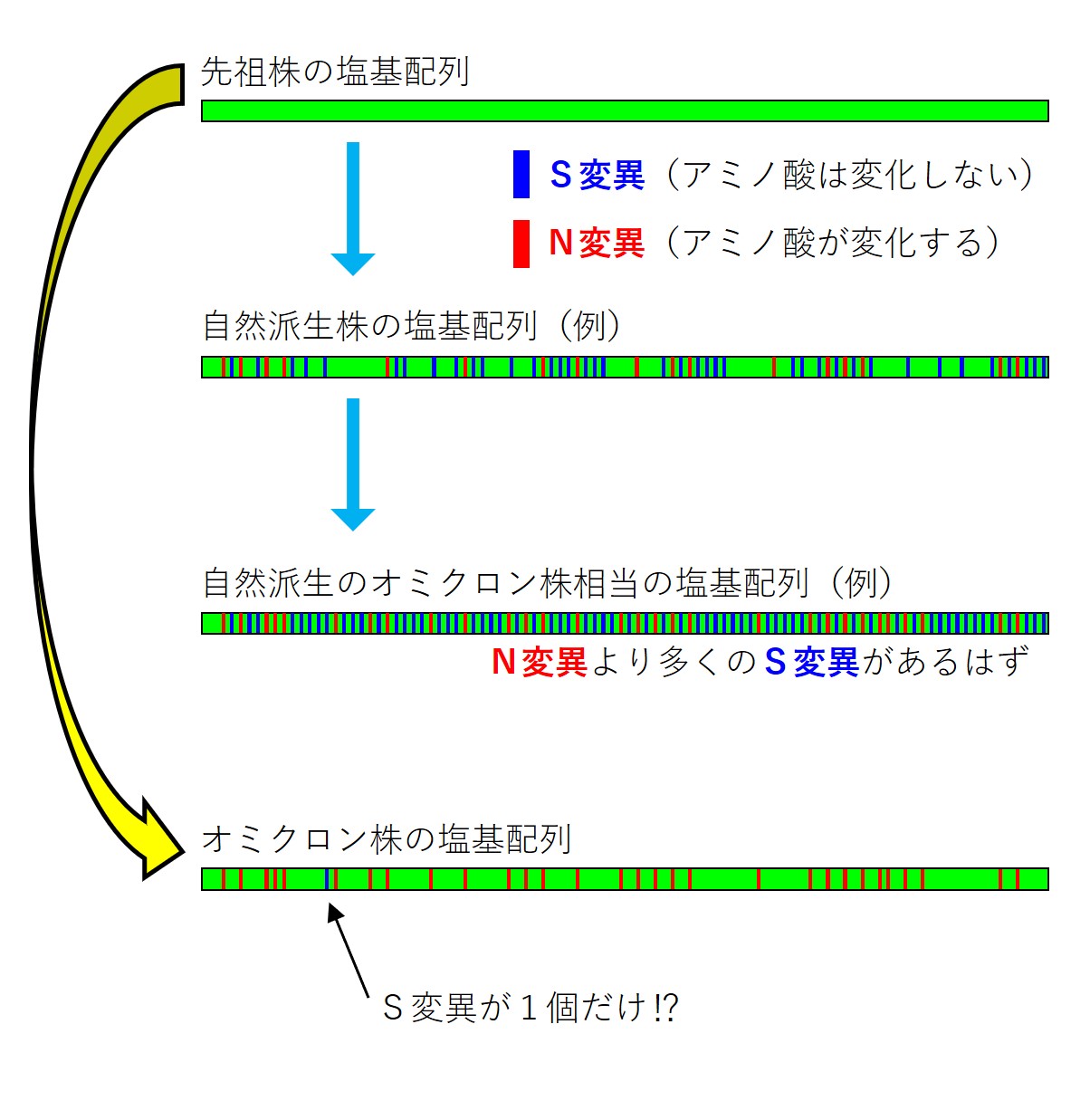
RNAの変異には アミノ酸が変化しない変異(S変異)と、アミノ酸が変化する変異(N変異)とがあります。
アミノ酸が変化しないS変異(=ウイルスタンパクが変化しない変異)は、ウイルスの各種能力には影響しません。そのようなS変異はウイルスの増殖に対して有利にも不利にも働かないので、増えも減りもせず、そのまま変異として残ります。
しかし、N変異でアミノ酸が変化すると、ウイルスタンパクの構造や機能が変化して、どちらかというと多くの場合ウイルスの増殖には不利になります。そのため、そのようなN変異は子孫には広まらないで淘汰される(消えていく)ことになります。
まれにウイルスの増殖に有利となるようなN変異がおこると、その変異はウイルスの増殖にともなって広まっていきます。
ウイルスの増殖に有利になるN変異と一緒になったS変異は、N変異と一緒に増えて広まります。逆に、ウイルス増殖に不利になるN変異と一緒になったS変異は、一緒に淘汰されて消滅します。
ウイルスのRNAでは、こうした変異が次々に現れては消えています。
まったくランダムに1塩基の置き換えが起こる場合、S変異の何十倍もN変異の方がたくさん起こりやすいのですが、上記の性質があるため、ウイルスが自然に増殖して世代交代していくとき、一般的には「アミノ酸が変化する変異(N変異)」より多くの「アミノ酸が変化しない変異(S変異)」が蓄積していきます。
しかし、2022年1月に日本での流行が始まった新型コロナウイルスのオミクロン株の塩基配列と、その先祖である武漢株の塩基配列を比較すると、アミノ酸が変化する変異(N変異)30個に対して、アミノ酸が変化しない変異(S変異)は1個しか認められなく、武漢株の自然な変化で(自然な変異の蓄積で)オミクロン株が派生したとはとても考えられない(下図)、というのが荒川央さんの分析結果です。
次は、人工ウイルスの証拠をもう少し詳しく説明します。
タンパクの基本骨格は、約20種類のアミノ酸が一列に繋がった鎖のようなものです。アミノ酸が一列につながったものを ペプチド と呼び、たくさんつながったものを ポリペプチド と呼びます。タンパクの基本骨格はポリペプチドです。
「約20種類のアミノ酸が一列に繋がった鎖のようなもの(ポリペプチド)」が、タンパクの基本骨格となります。
この長い線状の基本骨格が、くねくねとねじれたり、折れ曲がったりして、内部に特別な金属イオンを包み込んだり、基本骨格の一部に糖鎖(ブドウ糖のようなものが鎖のようにつながったもの)をくっつけたりして出来上がったのがタンパクの完成品です。
【参考】 立体構造のイメージをつかみたいとき →→→ 「タンパク質の立体構造」 という画像をクリックすると動画による説明が始まります(1分21秒)。
さて、このタンパクの基本骨格(ポリペプチド)のアミノ酸配列を決めているのが 遺伝子 と呼んでいるものです。遺伝子はタンパクのアミノ酸配列の情報を持っています。
遺伝子がどのような方法でアミノ酸配列の情報を持っているのかを理解できれば、荒川央さんの記事を読むことができるようになります。
遺伝子は、DNA 又は RNA の形で存在します。
DNA や RNA は、4種類の ヌクレオチド が一列に並んでできています。
約20種類のアミノ酸が一列に繋がった鎖のようなものがタンパクの基本骨格でしたが、4種類のヌクレオチドが一列に繋がった鎖のようなものがDNAやRNAです。
1つのヌクレオチドは、<リン酸、五炭糖、塩基>で構成されています。
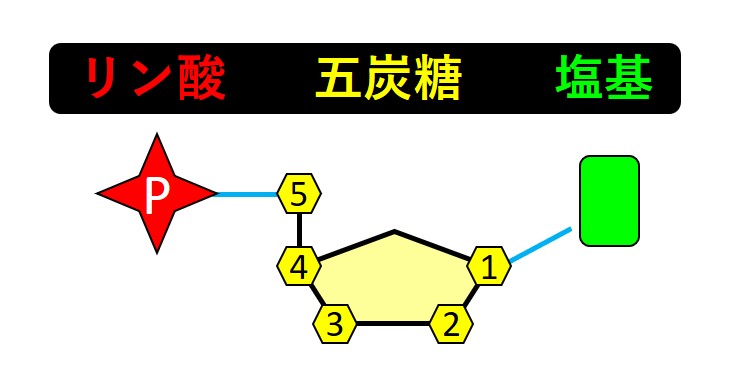
DNAの五炭糖はデオキシリボース、RNAの五炭糖はリボースという違いがあります。
DNAの塩基は4種類あり(A:アデニン、G:グアニン、T:チミン、C:シトシン)、そのどれか1つがDNAのヌクレオチドの塩基となります。
RNAの塩基も4種類あります(A、G、U:ウラシル、C)。DNAの T が、RNAでは U に置き換わっているだけです。
| ヌクレオチド | DNA | RNA |
| リン酸 | リン酸 | リン酸 |
| 五炭糖 | デオキシリボース | リボース |
| 塩基 | A | A |
| G | G | |
| T | U | |
| C | C |
DNAやRNAレベルで起こる変異のことを理解できるように、ひと通りの説明をしますが、覚えなければいけない要点は 「 DNAもRNAも4種類の塩基で区別されており、その4種類の塩基の配列順序がタンパクの基本骨格であるアミノ酸配列の順序を決めている 」 ということだけです。 五炭糖の名前も、塩基の名前も、DNAやRNAの構造も覚えておく必要はありません。
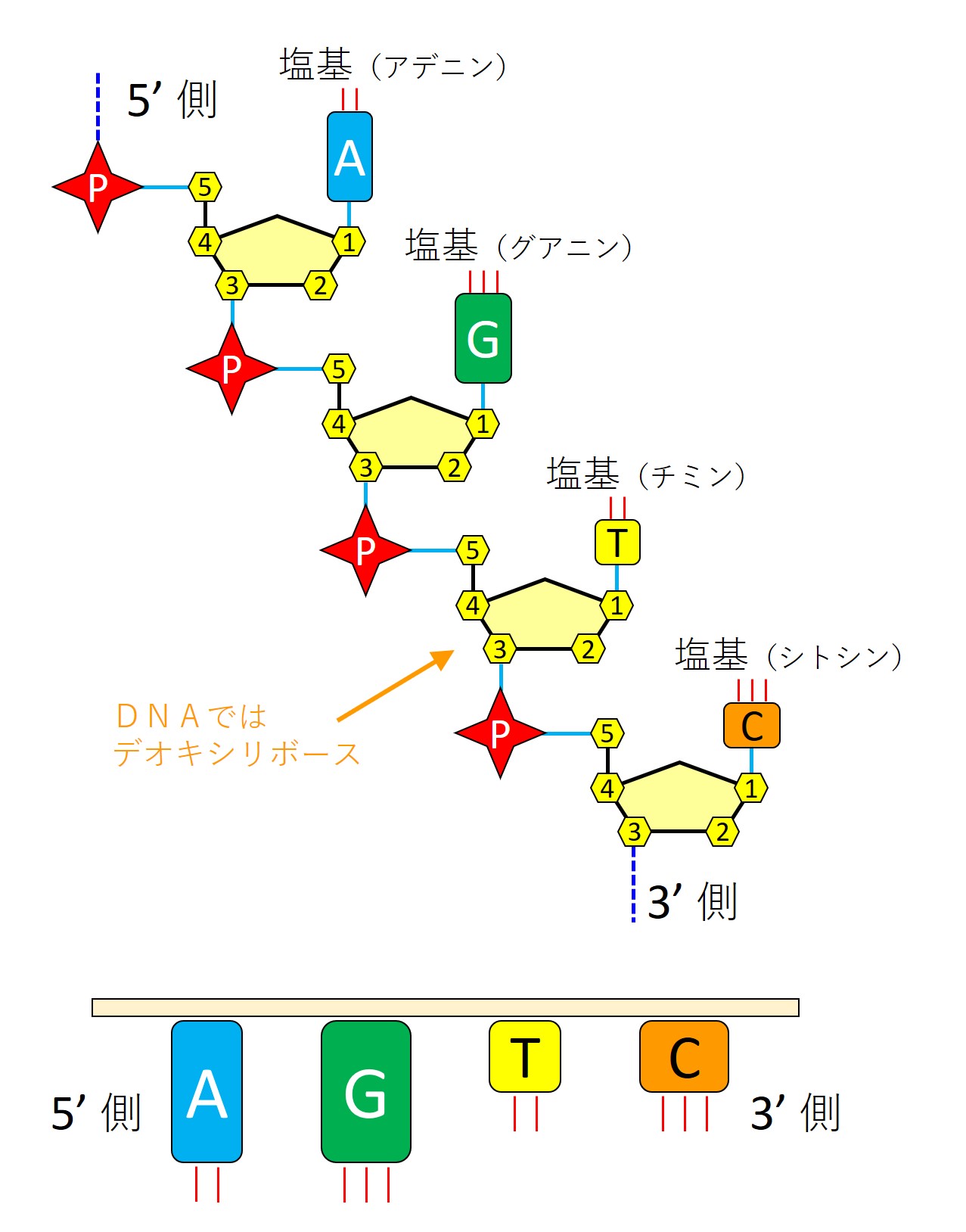
次図はDNAの例ですが、「P」がリン酸、「5個の炭素に1~5と番号を振ったもの」が五炭糖(デオキシリボース)、「A、G、T、C」が4種類の塩基です。ヌクレオチドが4個連なった例です。
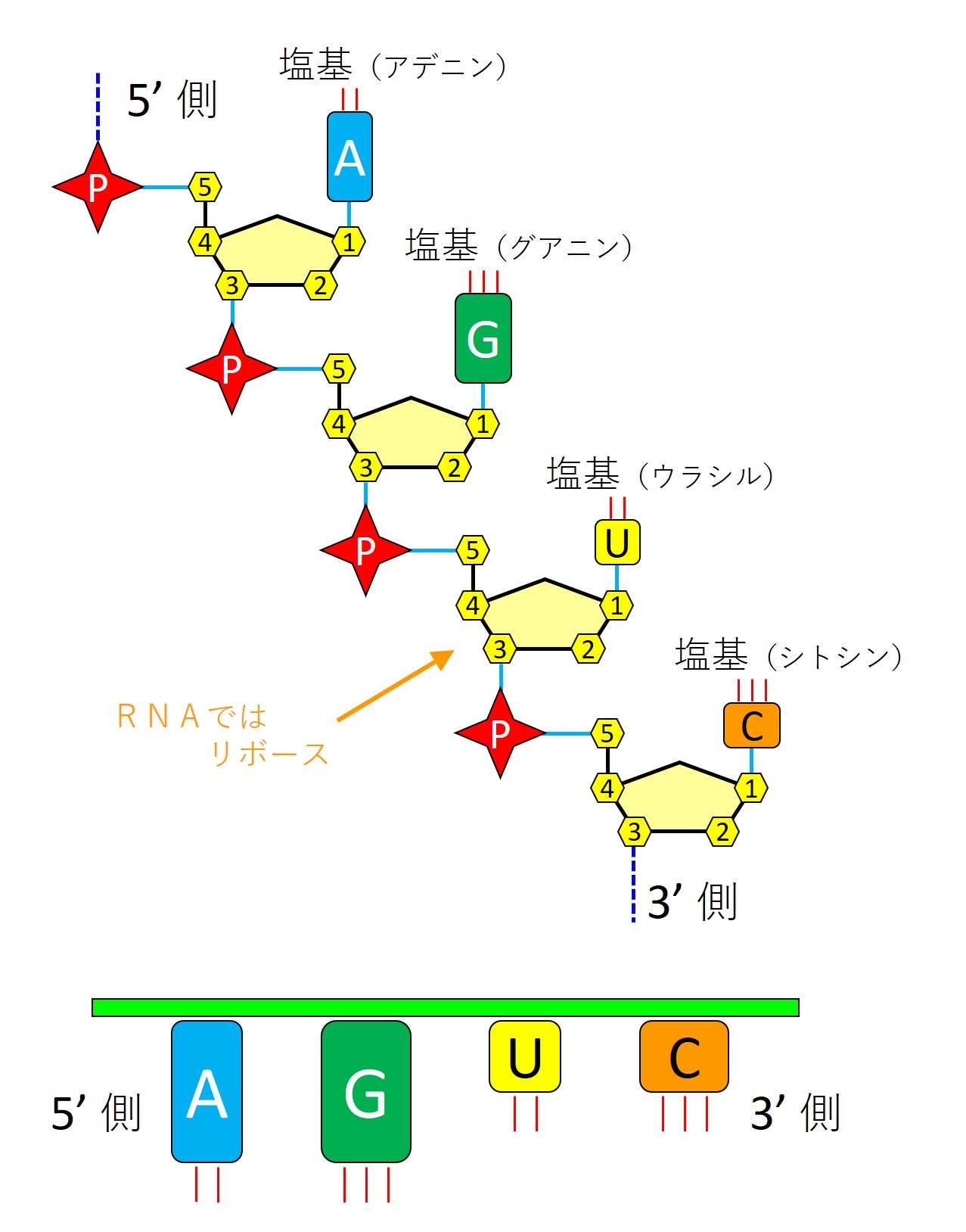
RNAの場合(次図)、五炭糖はリボース、4種類の塩基は「A、G、U、C」となります。DNAのチミン(T)は、RNAではウラシル(U)に替わります。
DNAやRNAの鎖には向きがあり、五炭糖の炭素番号を使って「5’側、3’側」を区別します。
4種類のヌクレオチドは、リン酸や五炭糖の部分は共通なので、先図の下に示すように省略し、塩基配列のみで表示します。先図の例では「5’→3’に向かって、AGTC(RNAではAGUC)」という配列になります。
DNAやRNAの塩基配列には向きがあるので、「AG」と「GA」は区別されるとだけ覚えておきましょう。
DNAやRNAは、この4種類の塩基配列を使って細胞内で作るタンパクの基本骨格(約20種類のアミノ酸の配列)の情報を保持しています。
このとき、塩基1個にアミノ酸1個を対応させると、4種類の塩基1個ではアミノ酸4種類にしか対応できません。
塩基2個でアミノ酸1個に対応させる場合、4種類の塩基2個では16通りとなり、やはり約20種類のアミノ酸に対応できません。
塩基3個でアミノ酸1個に対応させる場合、4種類の塩基3個では64通りとなり、約20種類のアミノ酸に余裕で対応可能となります。
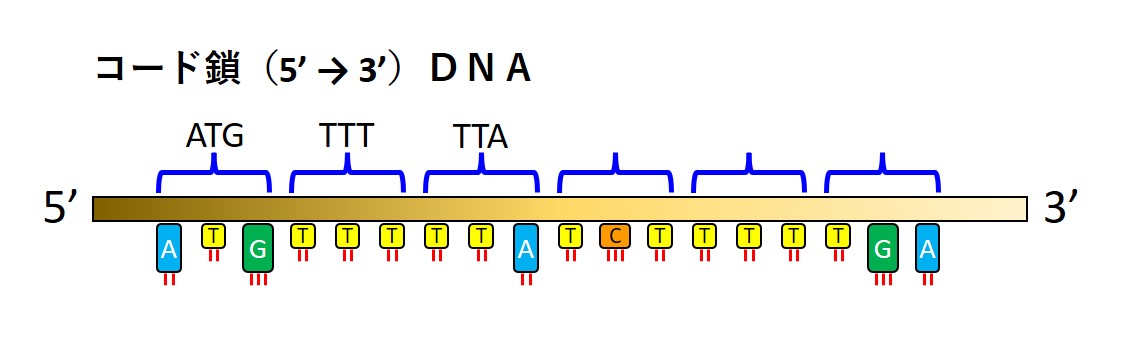
DNAでは、次図のように「5’側」から塩基3個ずつの配列がアミノ酸に対応しています。この3個の塩基セットを強調したいときは、特に「 コドン 」と呼びます。
このとき「ATG」という配列は メチオニン(Met)というアミノ酸に対応しており、「塩基配列(遺伝情報)読み取りの開始コドン」という特別な役割を担っています。
たとえば、次図の塩基配列;
「5' ATGTTTTTATCTTTTTGA・・・ 3'」は、
「5' ATG、TTT、TTA、TCT、TTT、TGA、・・・ 3'」と3塩基ごとに区切って読み取ることになります。
ヒトの遺伝情報(人体を構成する様々なタンパクの基本骨格を決めるアミノ酸配列の情報)の多くは細胞核の中で 二本鎖DNA の形で保管されています。二本鎖DNAは一本鎖DNAよりも「より安定した構造」であり、保管に向いています。
二本鎖DNAは、次図のように「コード鎖(5’→3’)」と「鋳型鎖(3’→5’)」とから構成されています。
コード鎖と鋳型鎖の向かい合った塩基同士の組み合わせは「AとT」、「GとC」がより強く安定した結合を作ります。
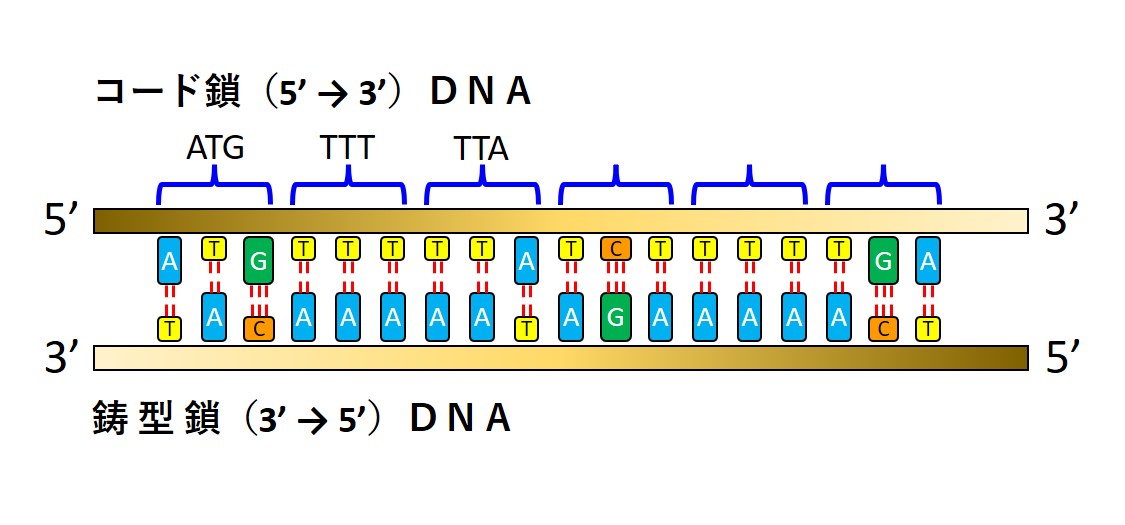
細胞がある特定のタンパクを作る時、二本鎖DNAをほどいてからコード鎖DNAの塩基配列を読む取る必要があります。
そのとき実際の読み取りは、コード鎖DNAではなく鋳型鎖DNAを逆読みする形で行われています(次図)。
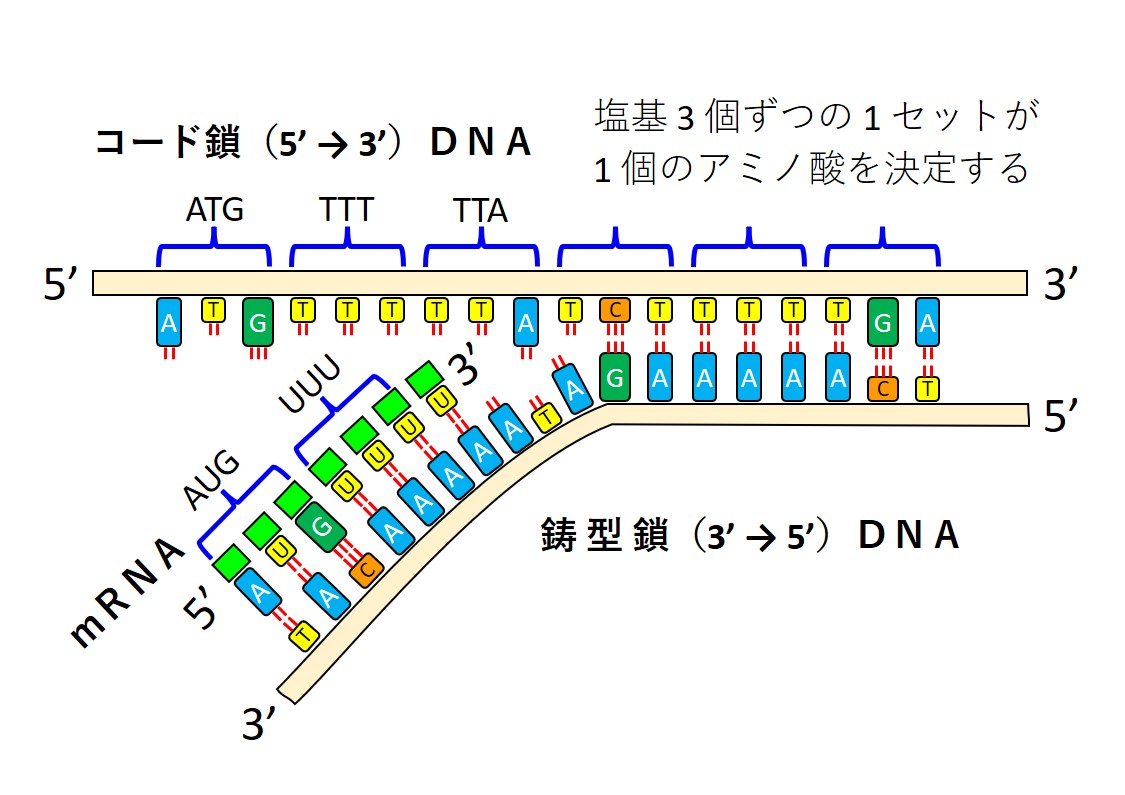
二本鎖DNAがほどけるとき、鋳型鎖DNAの「3’→5’」の向きに、RNAのヌクレオチドが1個1個くっついて連結していくと一本鎖RNAである mRNA(メッセンジャーRNA)が作られます。
DNAの4種類の塩基「A、G、T、C」は、RNAでは「A、G、U、C」となります。
そして「A=T」、「A=U」、「G≡C」の組み合わせが、強く結合する相補的塩基対となります。
先図では 5’→3’コード鎖DNAの「ATG」に対応するのは、3’→5’鋳型鎖DNAの「TAC」です。この「TAC」に対して、mRNAは「AUG」という塩基配列を作ります。RNAの「AUG」はDNAの「ATG」に相当します。
mRNAは、鋳型鎖DNAを使って作られるので、その塩基配列はコード鎖DNAに相当するものになっています(TがUに変化しているだけ)。
mRNAは細胞核の外に出て リボゾーム という場所に移動します。リボゾームでは、次図のように tRNA(トランスファーRNA) と呼ばれるものが、mRNA上のコドン(塩基3個のセット)に相当するアミノ酸を運んできます。
リボゾームでは、運ばれてきたアミノ酸が連結されてタンパクの基本骨格が作られます。
mRNAの(5’→3’)塩基配列の順序でアミノ酸は繋がっていきます。
mRNAの塩基配列はコード鎖DNAの(5’→3’)塩基配列の 写し となっています。
「DNA:塩基列 → RNA(mRNA:塩基列)」の情報の流れを 転写 と呼んでいます。「塩基列 → 塩基列」と写し取っているだけです。
「mRNA:塩基列 → tRNA(コドン→アミノ酸)の列:アミノ酸列」の情報の流れを 翻訳 と呼んでいます。「塩基列 → アミノ酸列」と変換しています。
「DNA → RNA → アミノ酸列」という情報の流れで、タンパクの基本骨格(アミノ酸が多数つながったポリペプチド)が作られます。
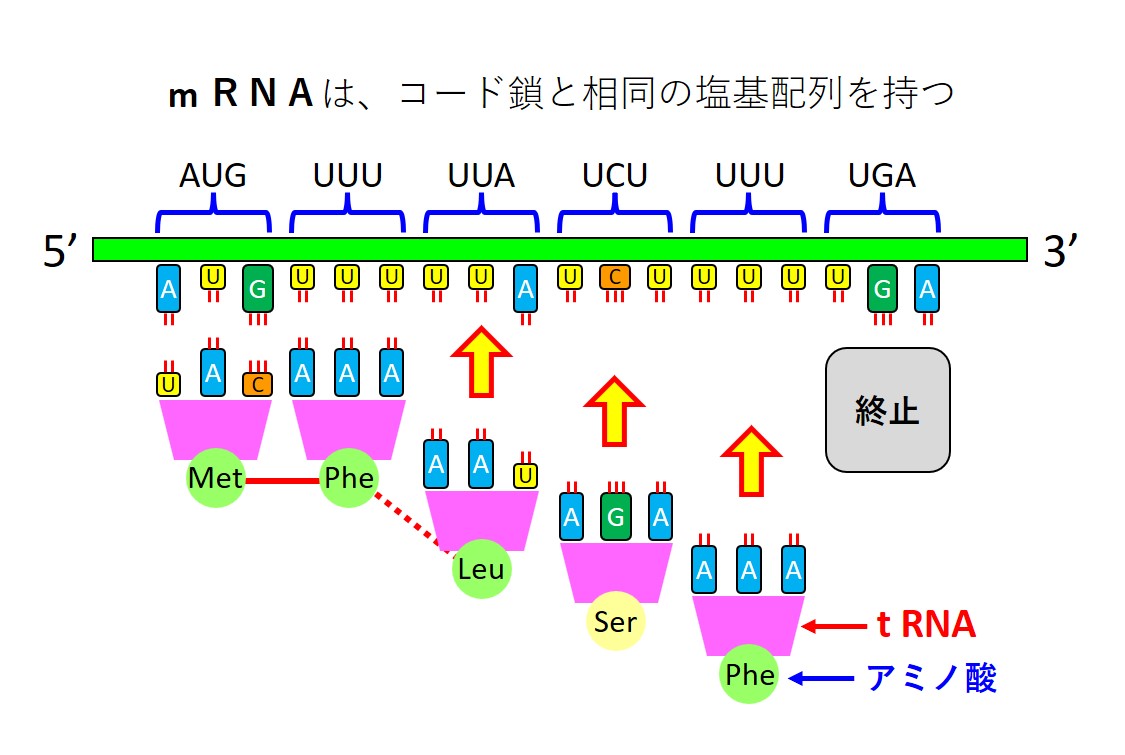
ここでは面倒な仕組みを理解して覚える必要はありません。DNAやRNAの塩基配列が遺伝情報を持っており、塩基3個ずつのセットが1個のアミノ酸を決めることだけ理解し覚えておけば十分です。
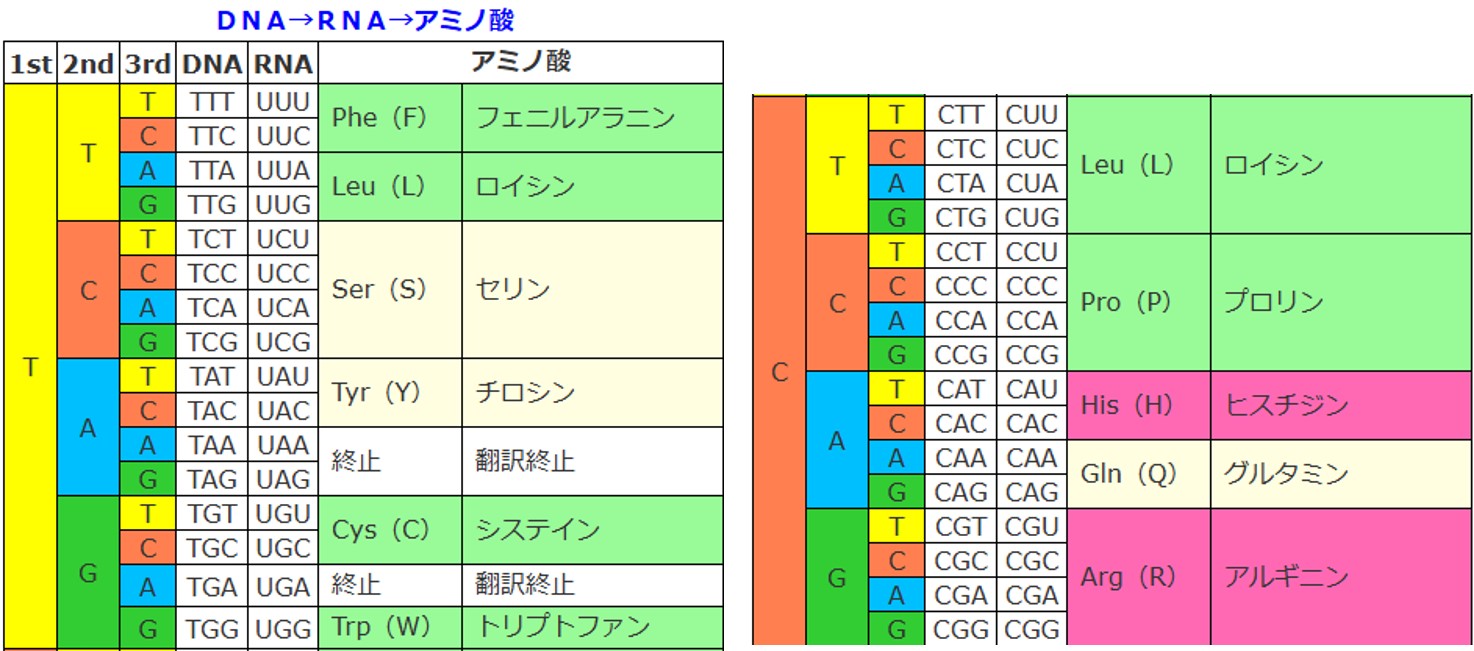
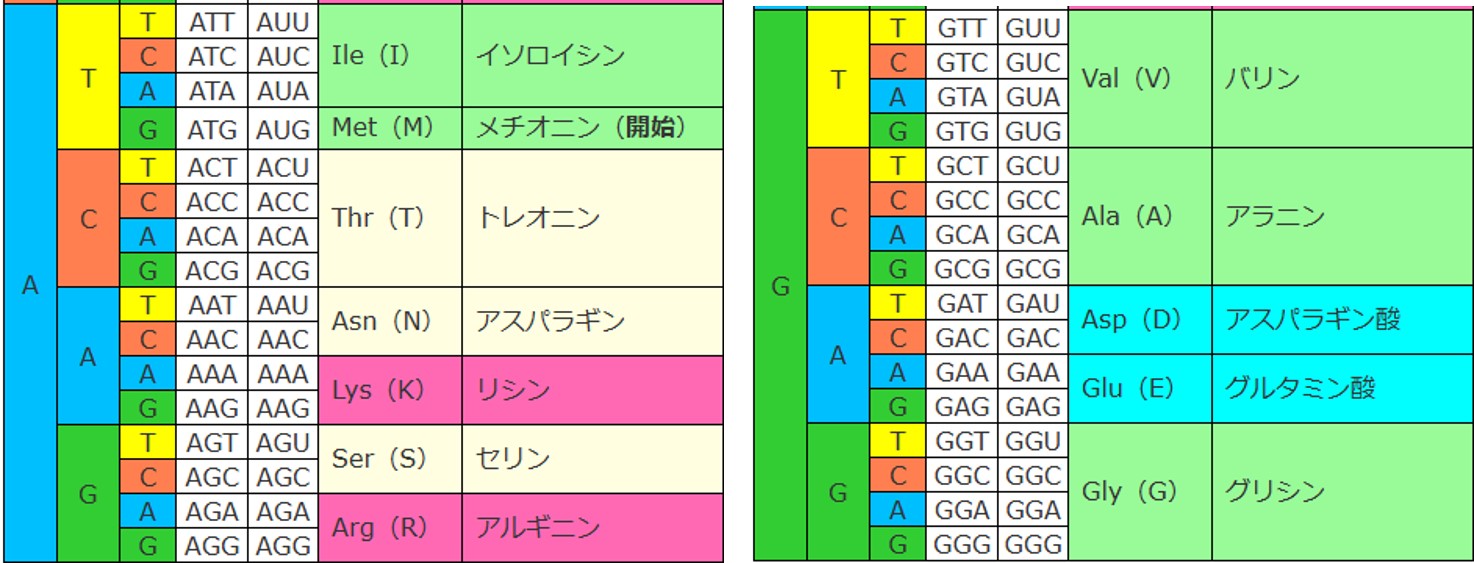
次の表は コドン表 と呼ばれています。DNA(およびRNA)の「5’→3’ 3個の塩基配列」が持つアミノ酸情報を表しています。
| 1st | 2nd | 3rd | DNA | RNA | アミノ酸 | |
|---|---|---|---|---|---|---|
| T | T | T | TTT | UUU | Phe(F) | フェニルアラニン |
| C | TTC | UUC | ||||
| A | TTA | UUA | Leu(L) | ロイシン | ||
| G | TTG | UUG | ||||
| C | T | TCT | UCU | Ser(S) | セリン | |
| C | TCC | UCC | ||||
| A | TCA | UCA | ||||
| G | TCG | UCG | ||||
| A | T | TAT | UAU | Tyr(Y) | チロシン | |
| C | TAC | UAC | ||||
| A | TAA | UAA | 終止 | 翻訳終止 | ||
| G | TAG | UAG | ||||
| G | T | TGT | UGU | Cys(C) | システイン | |
| C | TGC | UGC | ||||
| A | TGA | UGA | 終止 | 翻訳終止 | ||
| G | TGG | UGG | Trp(W) | トリプトファン | ||
| C | T | T | CTT | CUU | Leu(L) | ロイシン |
| C | CTC | CUC | ||||
| A | CTA | CUA | ||||
| G | CTG | CUG | ||||
| C | T | CCT | CCU | Pro(P) | プロリン | |
| C | CCC | CCC | ||||
| A | CCA | CCA | ||||
| G | CCG | CCG | ||||
| A | T | CAT | CAU | His(H) | ヒスチジン | |
| C | CAC | CAC | ||||
| A | CAA | CAA | Gln(Q) | グルタミン | ||
| G | CAG | CAG | ||||
| G | T | CGT | CGU | Arg(R) | アルギニン | |
| C | CGC | CGC | ||||
| A | CGA | CGA | ||||
| G | CGG | CGG | ||||
| A | T | T | ATT | AUU | Ile(I) | イソロイシン |
| C | ATC | AUC | ||||
| A | ATA | AUA | ||||
| G | ATG | AUG | Met(M) | メチオニン(開始) | ||
| C | T | ACT | ACU | Thr(T) | トレオニン | |
| C | ACC | ACC | ||||
| A | ACA | ACA | ||||
| G | ACG | ACG | ||||
| A | T | AAT | AAU | Asn(N) | アスパラギン | |
| C | AAC | AAC | ||||
| A | AAA | AAA | Lys(K) | リシン | ||
| G | AAG | AAG | ||||
| G | T | AGT | AGU | Ser(S) | セリン | |
| C | AGC | AGC | ||||
| A | AGA | AGA | Arg(R) | アルギニン | ||
| G | AGG | AGG | ||||
| G | T | T | GTT | GUU | Val(V) | バリン |
| C | GTC | GUC | ||||
| A | GTA | GUA | ||||
| G | GTG | GUG | ||||
| C | T | GCT | GCU | Ala(A) | アラニン | |
| C | GCC | GCC | ||||
| A | GCA | GCA | ||||
| G | GCG | GCG | ||||
| A | T | GAT | GAU | Asp(D) | アスパラギン酸 | |
| C | GAC | GAC | ||||
| A | GAA | GAA | Glu(E) | グルタミン酸 | ||
| G | GAG | GAG | ||||
| G | T | GGT | GGU | Gly(G) | グリシン | |
| C | GGC | GGC | ||||
| A | GGA | GGA | ||||
| G | GGG | GGG | ||||
さて、DNAやRNAの塩基配列は、様々な原因により常に変化しています。
ここでは、「性」の働きや「ウイルス」の働き等による(選択的で、多数の連続した)塩基の変化は対象としません。
ランダムに、非選択的に起こる塩基1個の他の塩基への変化を検討の対象とします。塩基1個なので 点突然変異(1塩基置換) と呼ばれています。
コロナウイルスは一本鎖RNAウイルスなので、以下ではRNAで説明します。
ウイルスを構成するタンパクの基本骨格のアミノ酸配列情報(遺伝子)は、ウイルスが持つRNA上の塩基配列に保持されています。
RNA上のすべての塩基は、ある一定の確率で点突然変異(1塩基置換)が起こっていると考えられます。実際には、RNA上の部位によって、また塩基の種類によって多少の偏りはあるでしょうが、ここでの考察で考慮する必要はありません。
このとき、点突然変異(1塩基置換)の影響を2段階で考えます。
まず、アミノ酸の変化が起こる変異かどうか です。
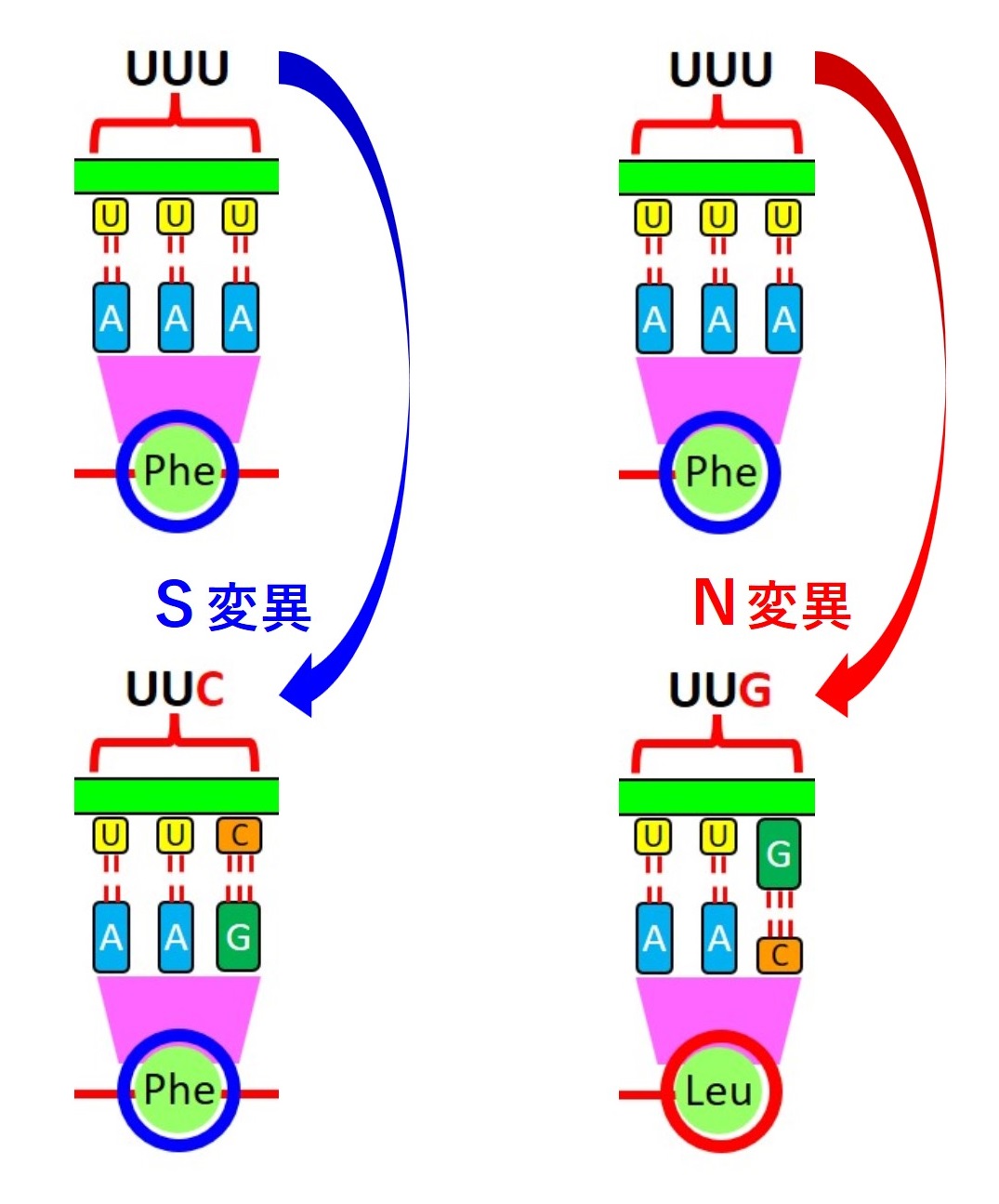
下図のように、RNAの「UUU」が「UUC」と変化しても、コドン表で見るとアミノ酸はフェニルアラニン(Phe)のままで変わりありません。
しかし、「UUU」が「UUG」と変化すると、アミノ酸はロイシン(Leu)に変わってしまいます。
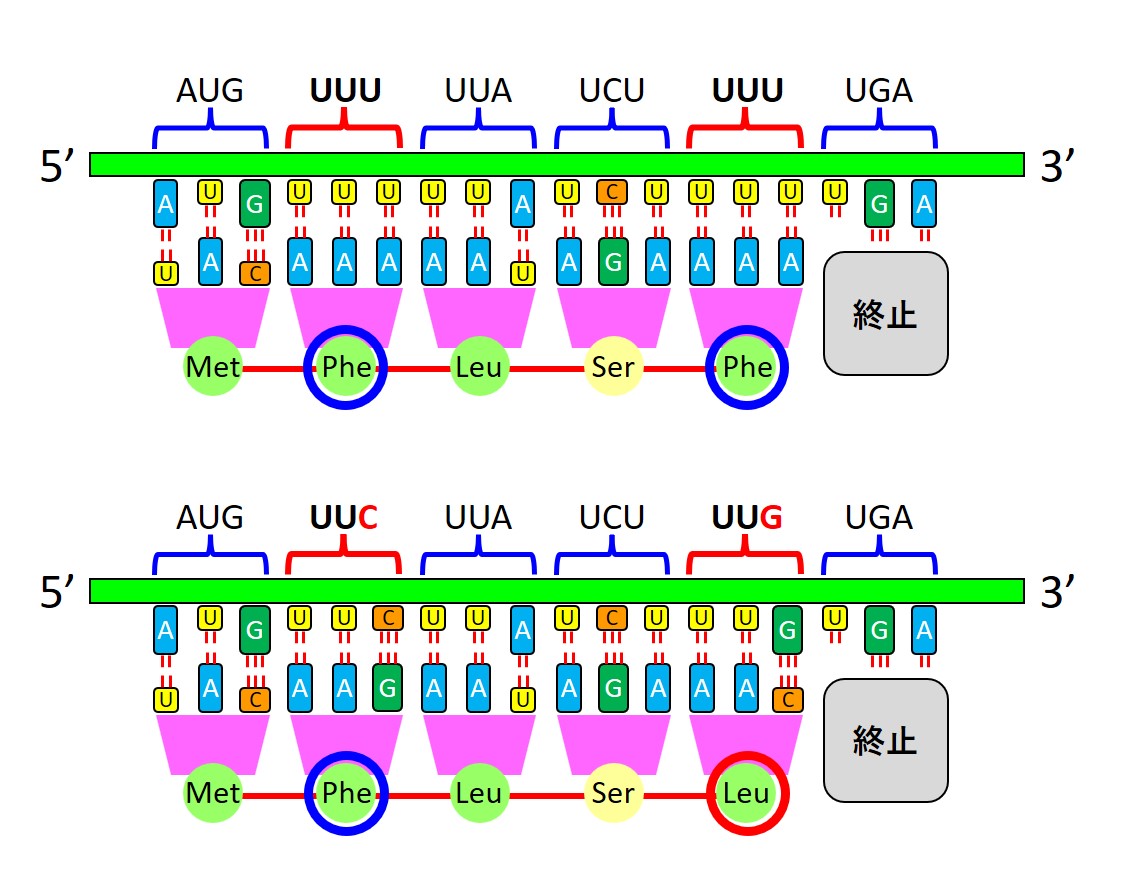
荒川央さんの説明文中では、前者(アミノ酸が変化しない塩基置換:同義置換)を「S変異」、後者(アミノ酸が変化する塩基置換:非同義置換)を「N変異」と呼んでいます。
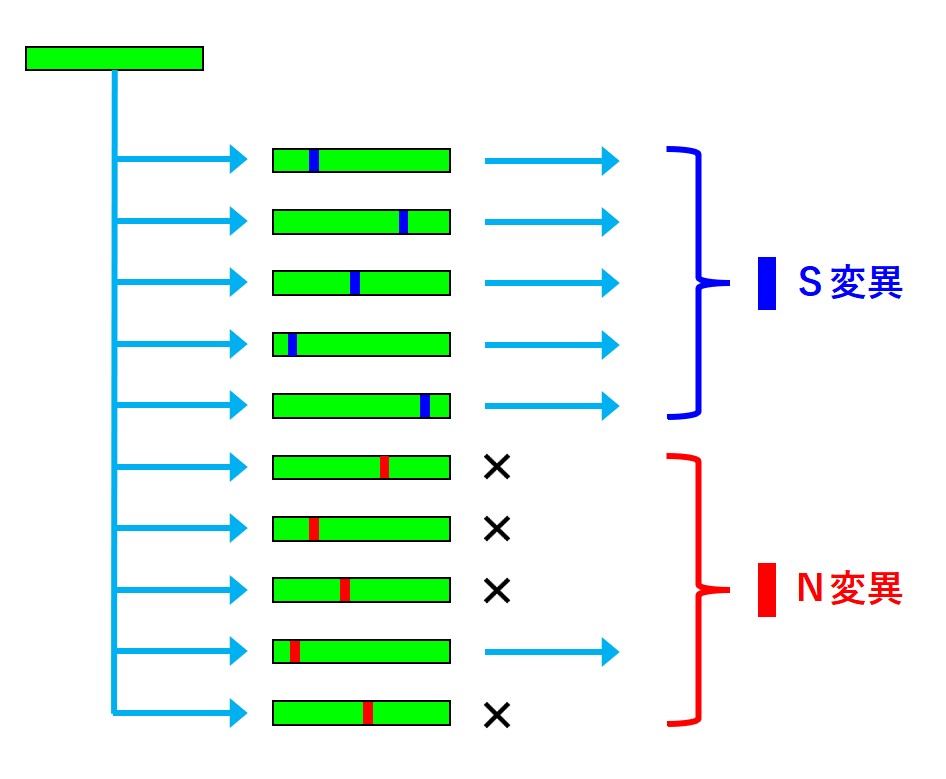
一般的に、塩基置換がほぼランダムで等確率に起こると仮定すると、S変異 の何倍もの N変異 が起こります。
例えば下表は、元の塩基配列「UUA(アミノ酸はロイシン)」に1塩基置換が起こる場合の変異例です。
全部で12通りありますが、「UUA」の1つ目の「U→U」、2つ目の「U→U」、3つ目の「A→A」は元々の塩基配列と同じなので省略しています。
| 1st | 2nd | 3rd | DNA | RNA | アミノ酸 | 変異型 | |
|---|---|---|---|---|---|---|---|
| T | T | T | TTT | UUU | Phe(F) | フェニルアラニン | N変異 |
| T | T | C | TTC | UUC | Phe(F) | フェニルアラニン | N変異 |
| T | T | A | TTA | UUA | Leu(L) | ロイシン | 元配列 |
| T | T | G | TTG | UUG | Leu(L) | ロイシン | S変異 |
| T | C | A | TCA | UCA | Ser(S) | セリン | N変異 |
| T | A | A | TAA | UAA | 終止 | 翻訳終止 | N変異 |
| T | G | A | TGA | UGA | 終止 | 翻訳終止 | N変異 |
| C | T | A | CTA | CUA | Leu(L) | ロイシン | S変異 |
| A | T | A | ATA | AUA | Ile(I) | イソロイシン | N変異 |
| G | T | A | GTA | GUA | Val(V) | バリン | N変異 |
次に、アミノ酸に変化が起こる場合(N変異の場合)、それがウイルスの流行拡大に有利に働くか不利に働くか です。
一般的に、長い進化の歴史を持つ重要なタンパクでは、アミノ酸の変化の大部分はウイルスの流行拡大に不利に働きます。つまり、そういう変異の多くは淘汰されて消えていきます。
同じことの言い換えとなりますが、ダメな変異の多くが消えることで、重要なタンパクが選び抜かれて残っているのです。
したがって、タンパクの構造や機能に大きな変化をもたらす変異は、(長い歴史を経た後では)たいていの場合、ウイルスの流行拡大には不利に働き、そういう変異を持つウイルスは消滅します。
N変異 の多くは、消えていく変異です。
しかし稀に、アミノ酸の変化がウイルスの流行拡大に成功をもたらした場合、そういうN変異は残り、増えていきます。
コロナウイルスのRNAでは、(ちょっとした偏りは無視すると)全塩基にほぼ一定の確率で点突然変異(1塩基置換)が起こっていると考えることができます。
アミノ酸に変化をもたらさない S変異 は、それが原因で淘汰されることはなく、そのまま残る と考えられます。
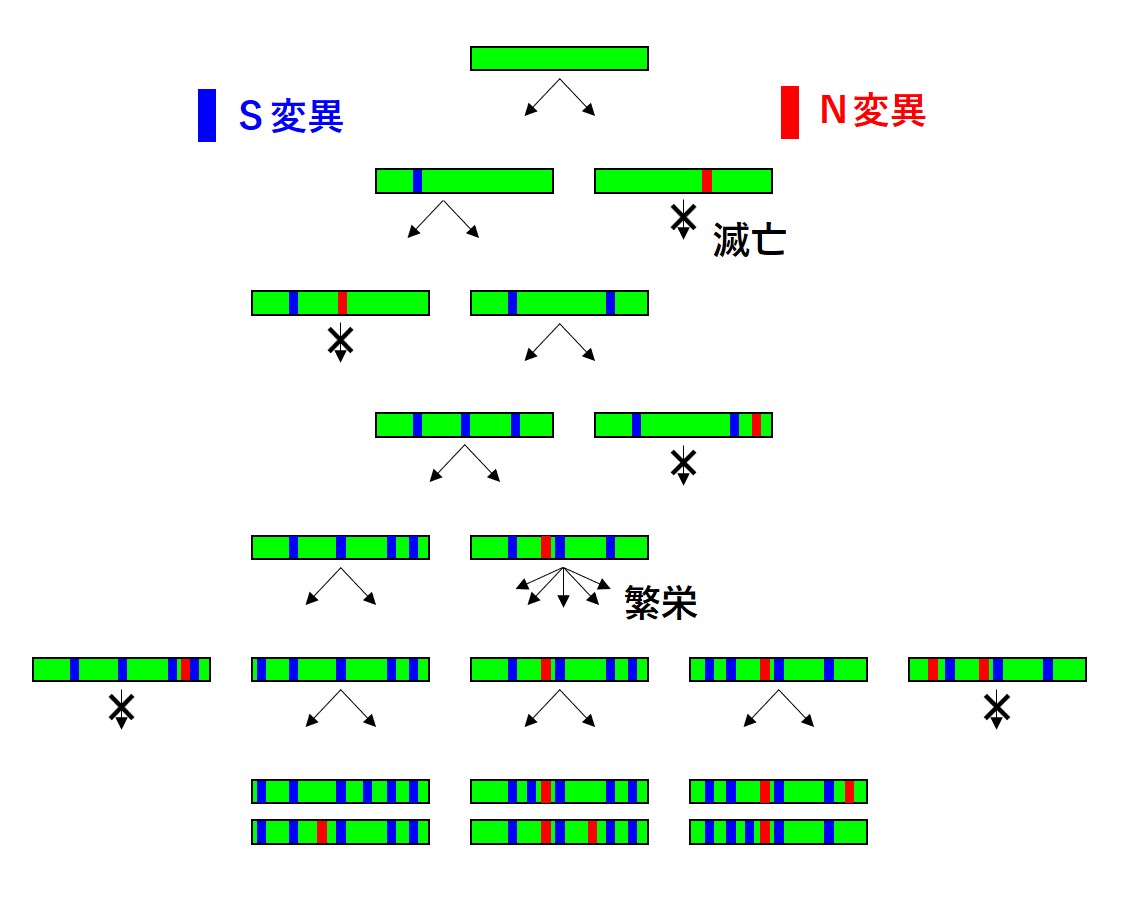
以上を整理すると、
「アミノ酸が変化しないS変異は、それが原因で増えも減りもせず、残る」
「アミノ酸が変化するN変異の大部分は淘汰されて消えるが、一部はウイルスの流行拡大に成功をもたらして劇的に増える」
「成功したN変異とたまたま一緒になったS変異は、一緒に増える」
「失敗したN変異とたまたま一緒になったS変異は、一緒に消滅する」
ということになります。
N変異はチャレンジャー(挑戦者)的、S変異は一緒になったN変異の運命に伴って受動的に増減します。
突然変異ではS変異よりもたくさんのN変異が起こるのですが、多くのN変異が淘汰で消えるので、多くの世代交代の後にはS変異のほうがたくさん残ることになります。
全体主義社会と同じです。出る杭は打たれる、打たれても出てくる杭は抜かれる。
イメージを描くと下図のようになります。
下図では、「ウイルスは増えるたびに変異が1回起きる」、「S変異とN変異の生じる比は1:1」、「N変異の 2/3 は増殖に不利となり消滅」、「N変異の 1/3 は増殖に有利となり繁殖」と、図の範囲内にいろいろな状況を描けるように仮定しています。
N変異をもっと細かく見ると、ウイルスの流行拡大にとって重要なアミノ酸配列の部分では、アミノ酸を変えるような点突然変異(1塩基置換)は淘汰されるので元々の塩基配列が温存されやすく、さほど重要でないアミノ酸配列の部分ではアミノ酸を変えるような点突然変異(1塩基置換)も淘汰されることなく残ることになります。
タンパクの場所によってN変異の持つ意味合いも異なってきますが、そこまで調べなくてもコロナウイルス(オミクロン株)の不自然さは明らかだったようです。
荒川央さんは、オミクロン株とその先祖株とのRNA塩基配列を比較しました。
ウイルスの自然な進化では、成功した1個のN変異に対して何倍ものS変異が伴っていて、ウイルス変異の多くの試行錯誤の結果だということがわかるのですが、荒川央さんが先祖株との比較対象としたオミクロン株では、N変異が30個もあるのに、S変異が1個しかなかったのです。

つまり、オミクロン株のN変異の大部分は人工的に加えられた変異であるとしか理解できないということです。
「DNA→RNA→タンパク」についてあまり知らなかった方も、直接記事を読んで内容を理解できるようになっていると思います。
プレプリント Mutation signature of SARS-CoV-2 variants raises questions to their natural origins. Arakawa, Hiroshi June 1, 2022
掛谷英紀(かけやひでき)さんの著書:「学者の暴走」(2021.07.02、扶桑社新書)
掛谷さんは、オミクロン出現以前から人工ウイルスの可能性を指摘されていました。
「学者の暴走」の目次
第1章 新型コロナウイルスと悪魔の科学
情報隠蔽がもたらしたパンデミック
武漢で行われていた危険な研究
陰謀論とのレッテルと戦う
あまりに危険な科学者たち
第2章 科学とは何か
第3章 日本の科学の弱点
第4章 世界の学問の危機
第5章 学問の再建に向けて
「コロナワクチンが危険な理由」、荒川央(あらかわひろし)、花伝社(税抜き¥1,500)、2022.03.25、「オミクロン変異考察」:192 ~ 203 ページ

「新型コロナは人工物か?」 宮沢孝幸(みやざわたかゆき) 2024.08.05 PHP新書

77 ~ 79 ページ:
(オミクロン変異体の「BA.1」は)非同義置換(N変異)が 30 箇所もあるのに同義置換(S変異)がわずか 1 箇所であることから、人工ウイルスであることは明白だと私は考えました。それでもなお、多くの人たちは、自然でもそのような変異は免疫不全の患者ではあり得ると主張しました。私は非同義置換に偏ることはあっても、ほとんどが非同義置換になるのはどんな状況でもあり得ないと考えています。
オミクロン変異体「BA.2」についても調べてみると、「BA.1」とまったく同じように、異常な変異が起こっていることがわかりました。「BA.2」のスパイクタンパクの配列を調べたところ、同義置換は「BA.1」と同じくわずか 1 箇所で、非同義置換は 24 箇所もあったのです(本の口絵図 0-1)。
オミクロン変異体がこのような異常な変異をしていることがわかったので、次に、他の変異体、アルファ、ベータ、ガンマ、デルタ、ラムダ、ミューについても同様に調べました。すると、その結果も驚くべきものでした。オミクロン程は変異していないものの、どれも同義置換はほとんどありませんでした。アルファとベータ、ガンマ、デルタ、ミューに至っては同義置換がゼロの分離株もありました(本の口絵図 0-1)。
さらに興味深いこともわかりました。
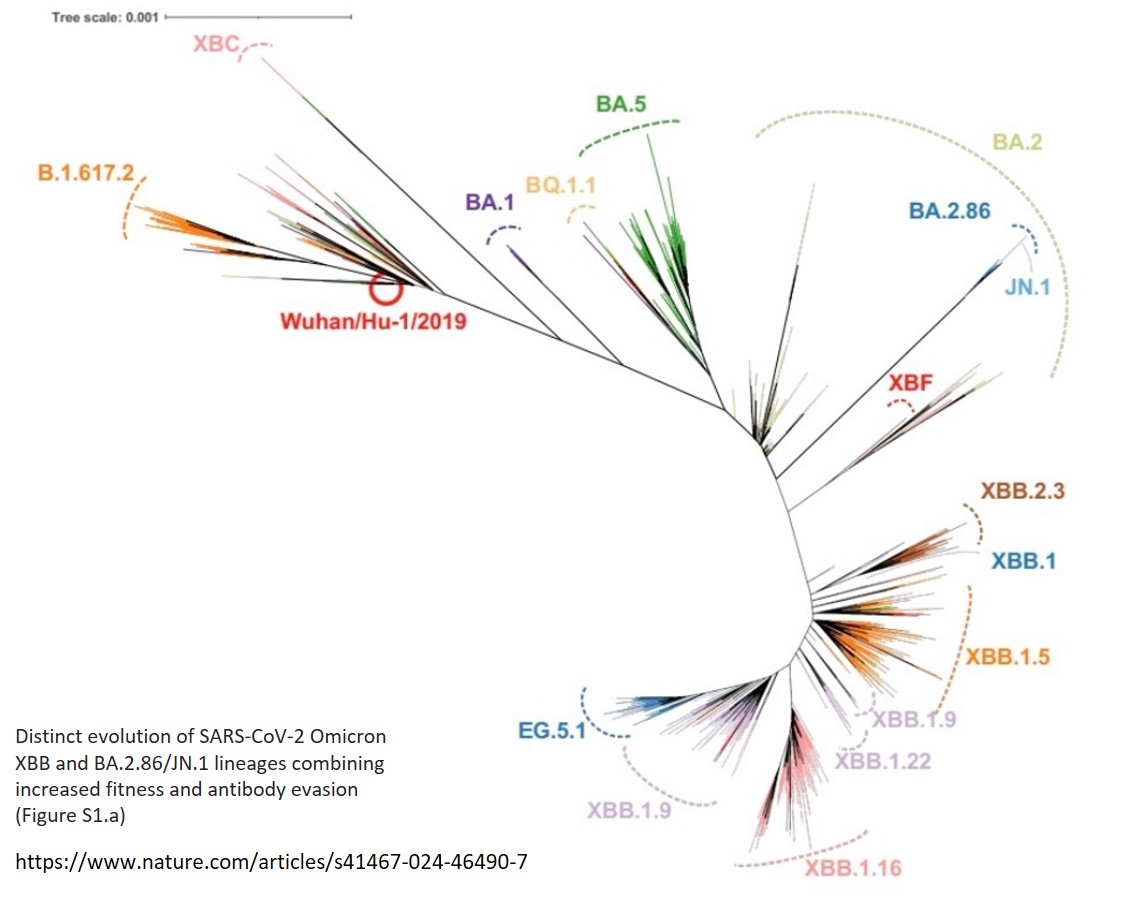
武漢株から時間経過とともにアルファ、ベータ、ガンマ、デルタ、ラムダ、ミューと変異体が出現したのですが、これらの変異にはまったく連続性が見られなかったのです(本の口絵図 0-1)。たとえば、アルファ変異体に変異がさらに蓄積していってベータ変異体やガンマ変異体になったというわけではなく、すべてが独立して武漢型から変異していたのです。しかし、出現した時間がずれているのです。
たとえ話・・・・(中略)・・・・実際には一部共通している変異もあるのですが、それぞれの変異体で変異の多くが新規のものだったのです。
新型コロナウイルスの新しい変異体が出現する時は、同義置換がほとんど入っていないのですが、その後の変異体の派生型では同義置換は入っていきます。不自然さはありません。この差異で生じた同義置換は時間経過とともに少しずつ蓄積していくのですが、新しい変異体が生じる時はそれがないのです。このようなことが何度も繰り返されたのです。
このおかしな現象に気付いた人は世界で何人もいました。国内ですと筑波大学の掛谷英紀(かけやひでき)先生、ミラノ在住の日本人研究者である荒川央(あらかわひろし)先生もそうです。掛谷先生も荒川先生のどちらもプレプリントサーバーに論文を投稿しています(Ref.8, 9)。私はそれを見て、さすがにこれはおかしいので大騒ぎになるだろうと考えていました。
私が2回目のコロナを発症したのは、令和5年2023年1月8日(日曜)です。
「3-5-2.コロナ(2回目)」を読むとき、ここを参照してください。
国立感染症研究所による「新型コロナウイルス ゲノムサーベイランスによる系統別検出状況:国内 新型コロナゲノムの PANGO lineage 変遷(2023/01/20 現在)」によると、(私が感染した)2023年第1週の時点で「BA.5 系」が 86.5 % となっています。
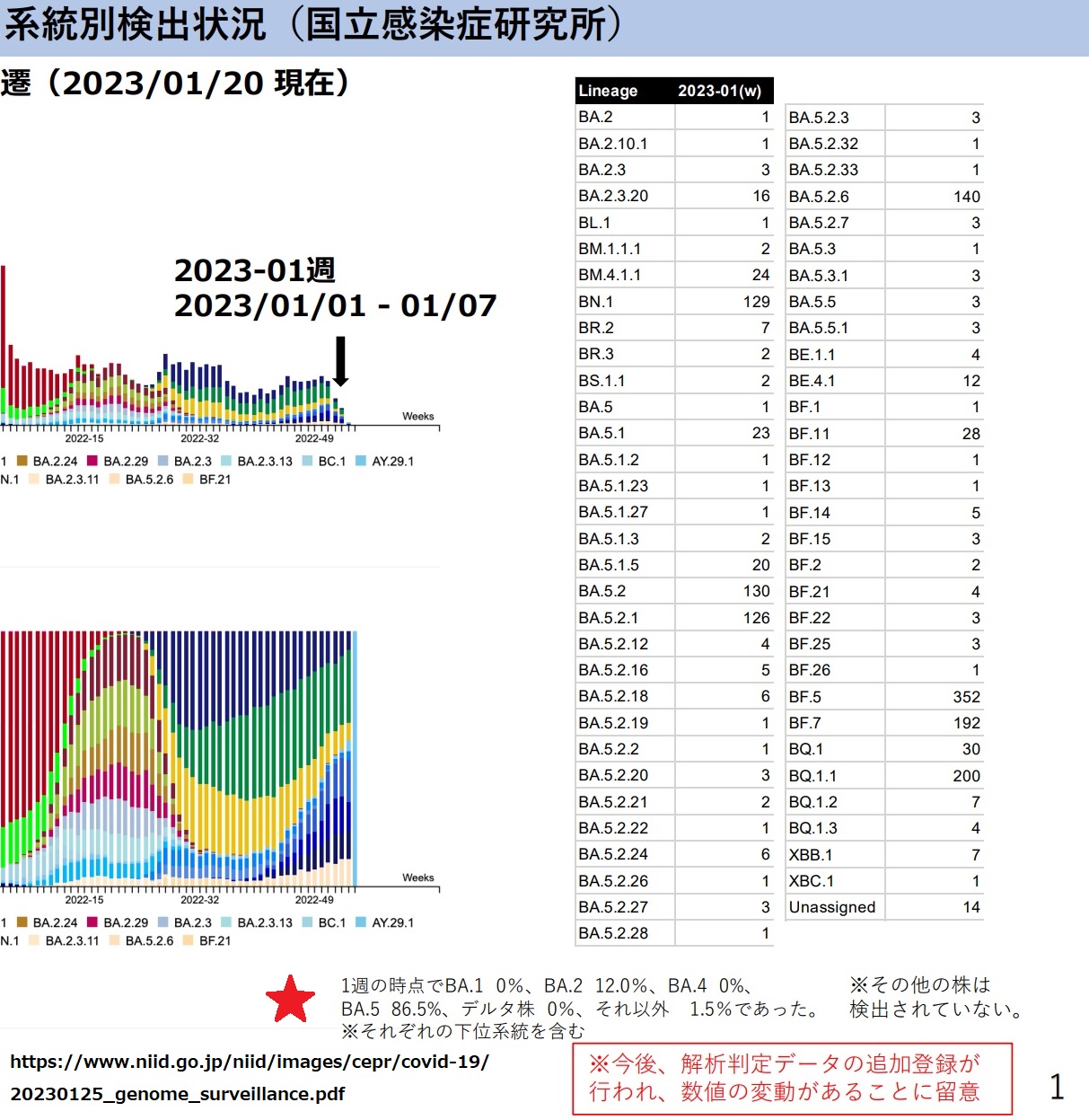
国立感染症研究所が報告した「民間検査機関の検体に基づくゲノムサーベイランスによる系統別検出状況:第1週 (2023年1月2日~2023年1月8日)」でも、日本における変異体の約9割が「BA.5 系」となっています。
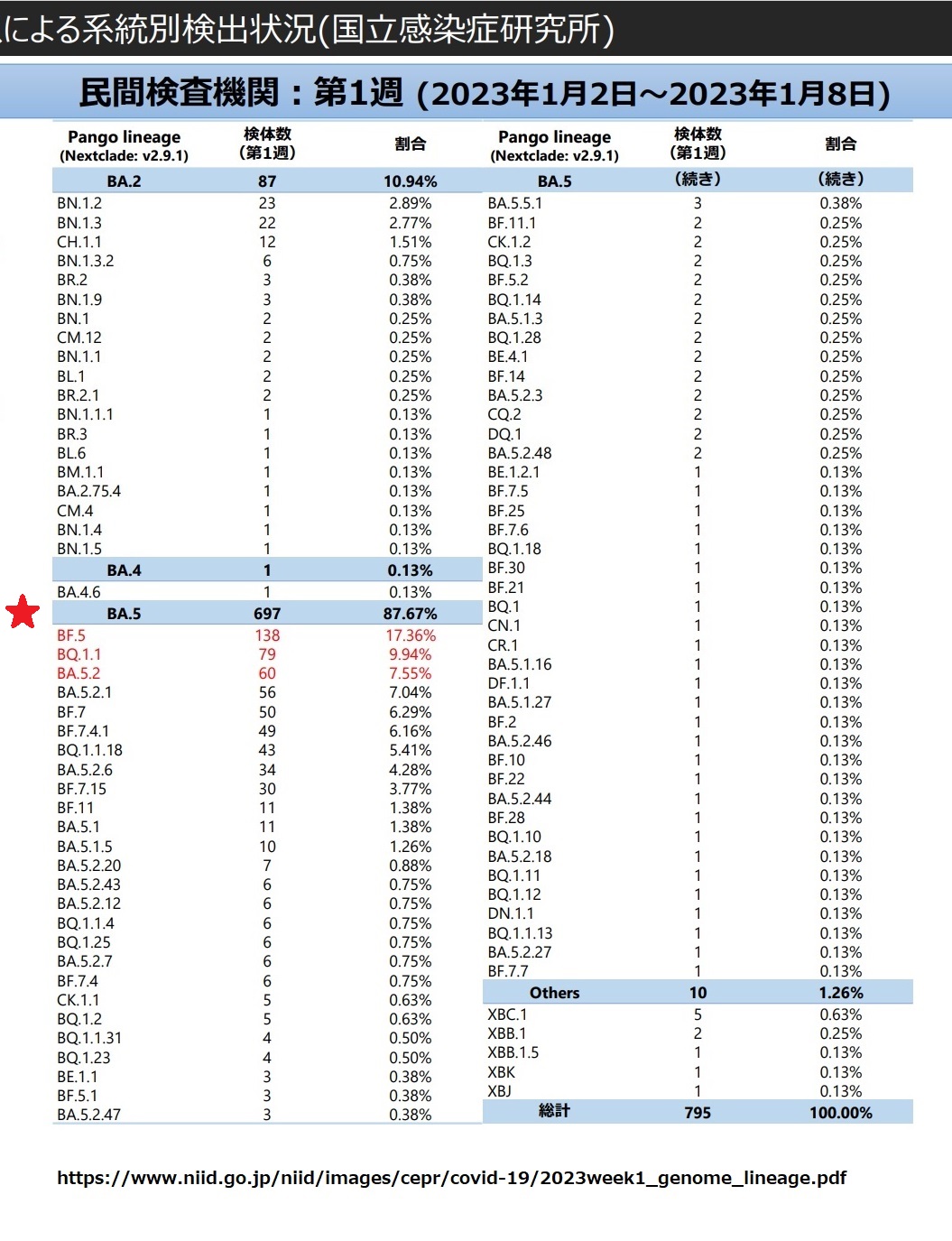
(参考:NIID(国立感染症研究所)の新型コロナウイルス感染症(COVID-19)関連情報ページ:▶ ▶ ▶)
都内居住、電車通勤で横浜勤務の私が1月初めに感染したコロナは、おそらく「BA.5 系」でしょう。
私が3回目のコロナを発症したのは、令和6年2024年1月28日(日曜)です。
「3-5-3.コロナ(3回目)」を読むとき、ここを参照してください。
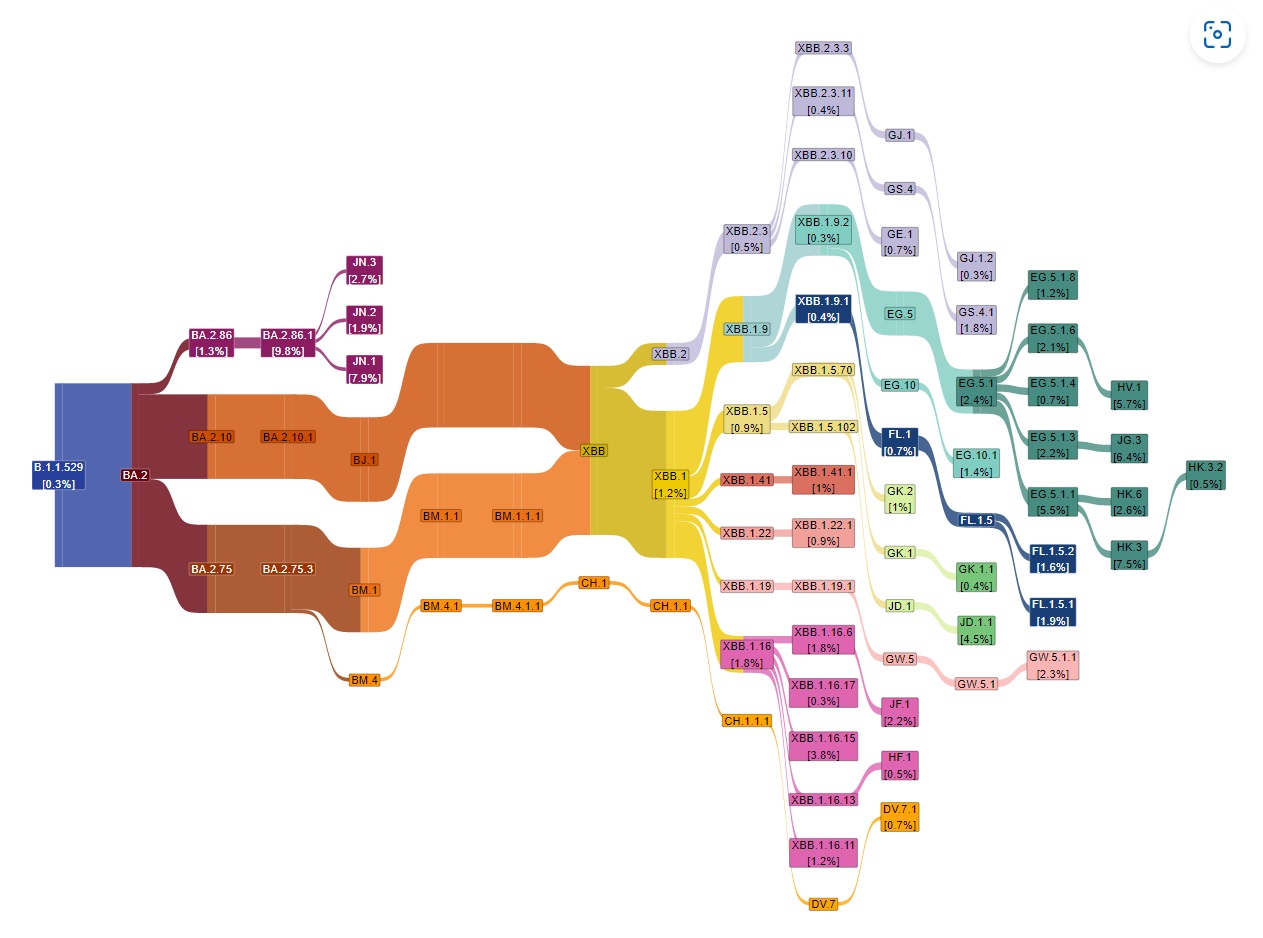
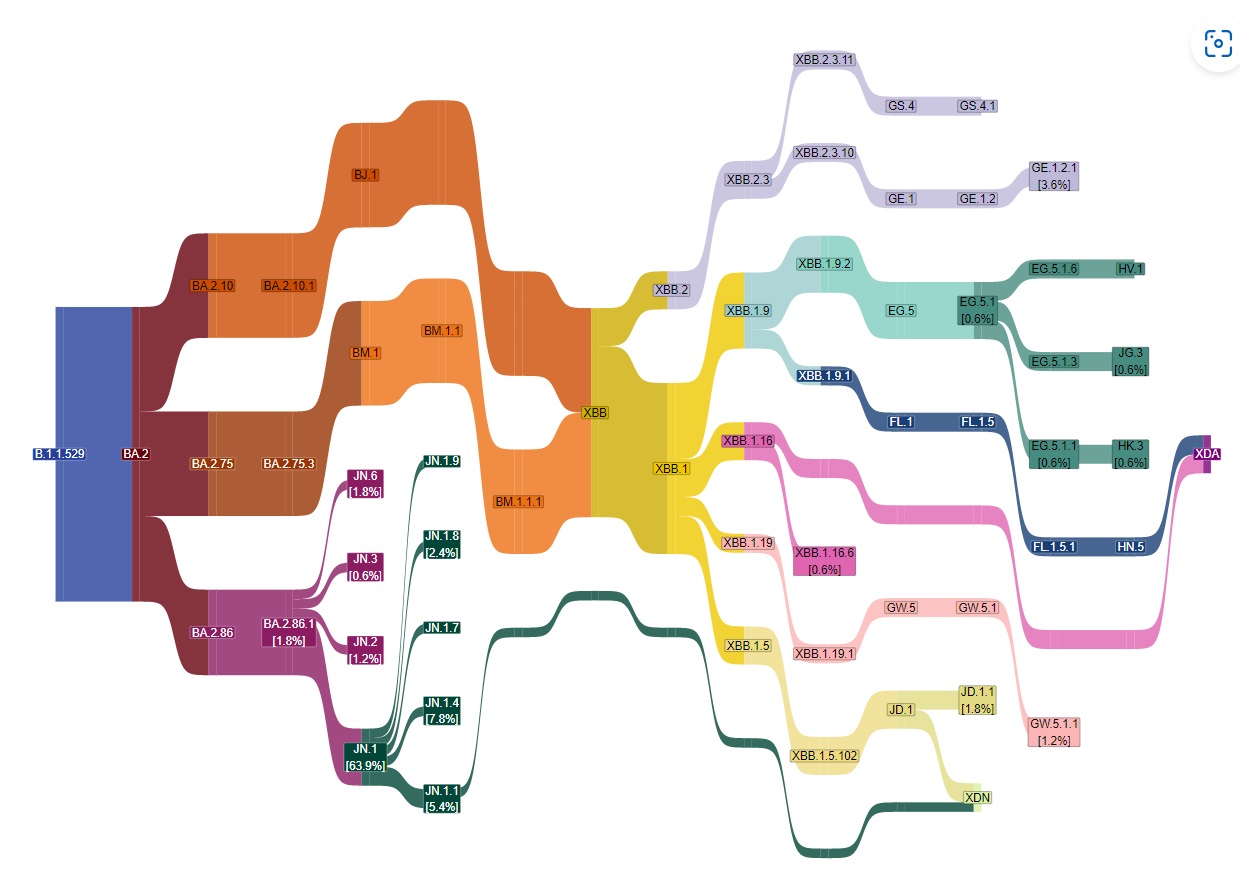
2023年の1月初めは「BA.5」が大流行していたので苦労しませんでしたが、2024年の1月は複数の変異系が流行しているのでしっかりと調べる必要がありました。
主流行系統は「BA.2系」でした。その中の「XBB.1系(少しずつ勢いが弱くなっている)」または「BA.2.86.1系(少しずつ勢いが強くなっている)」のどちらかである可能性が大きいと判断しました。
現在の流行系統:国立感染症研究所感染症疫学センター「新型コロナウイルス感染症サーベイランス週報:発生動向の状況把握:2024年第3週(1月15日~1月21日)」
多いのは「HK.3(XBB.1.9.2.5.1.1.3)」、「BA2.86.1(B1.1.529.2.86.1)」、「JN.1(B.1.1.529.2.86.1.1)」など
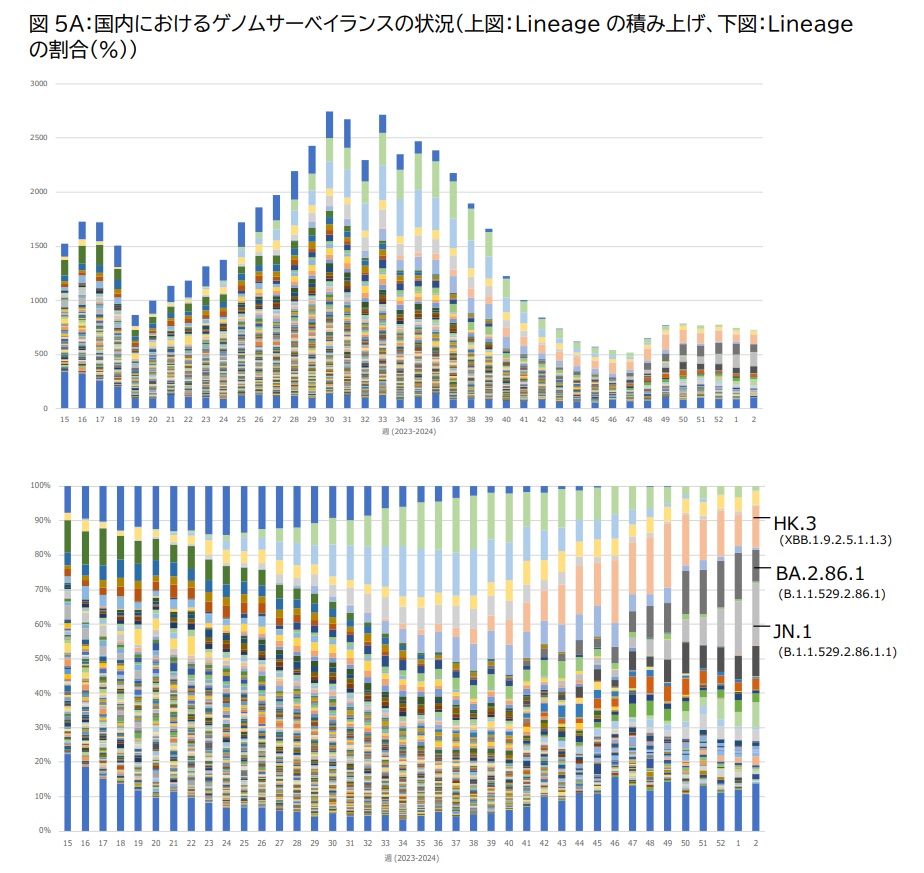
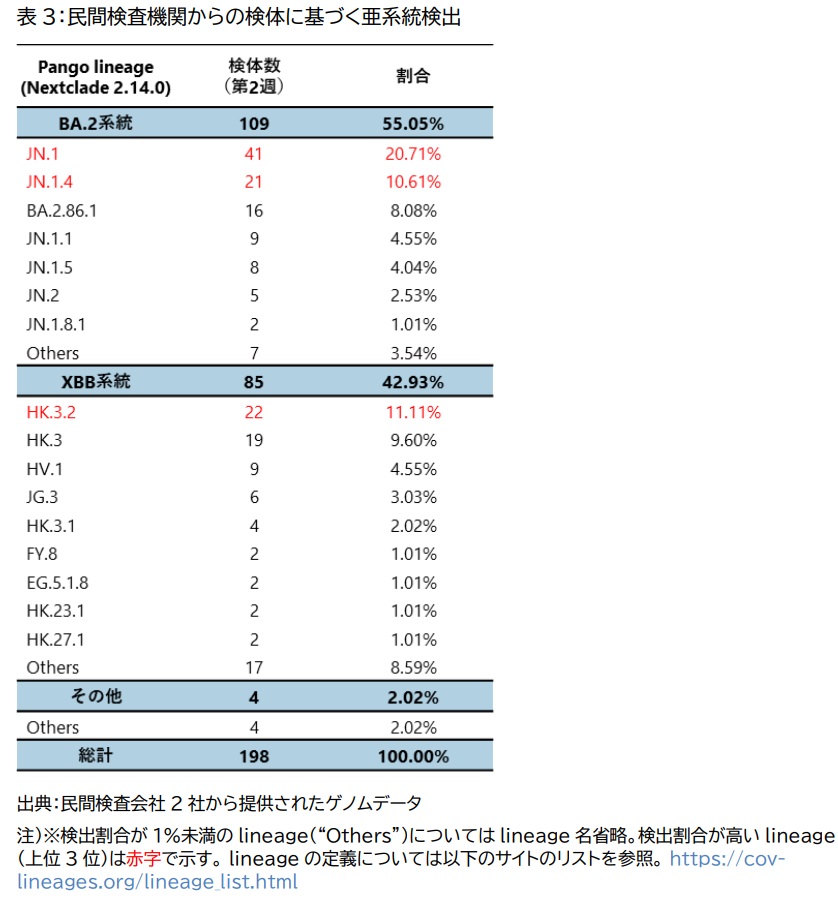
東京都の流行系統:東京都のモニタリング分析資料(令和6年2月22日公表):スライド9枚目:病原体サーベイランス(ゲノム解析)2024年令和6年(1月22日~2月4日)」
ほとんどが「BA.2系」であり、その5~8割が増加中の「BA.2.86系(JN.1など)」、5~2割が減少中の「XBB.1系(EG.5など)」
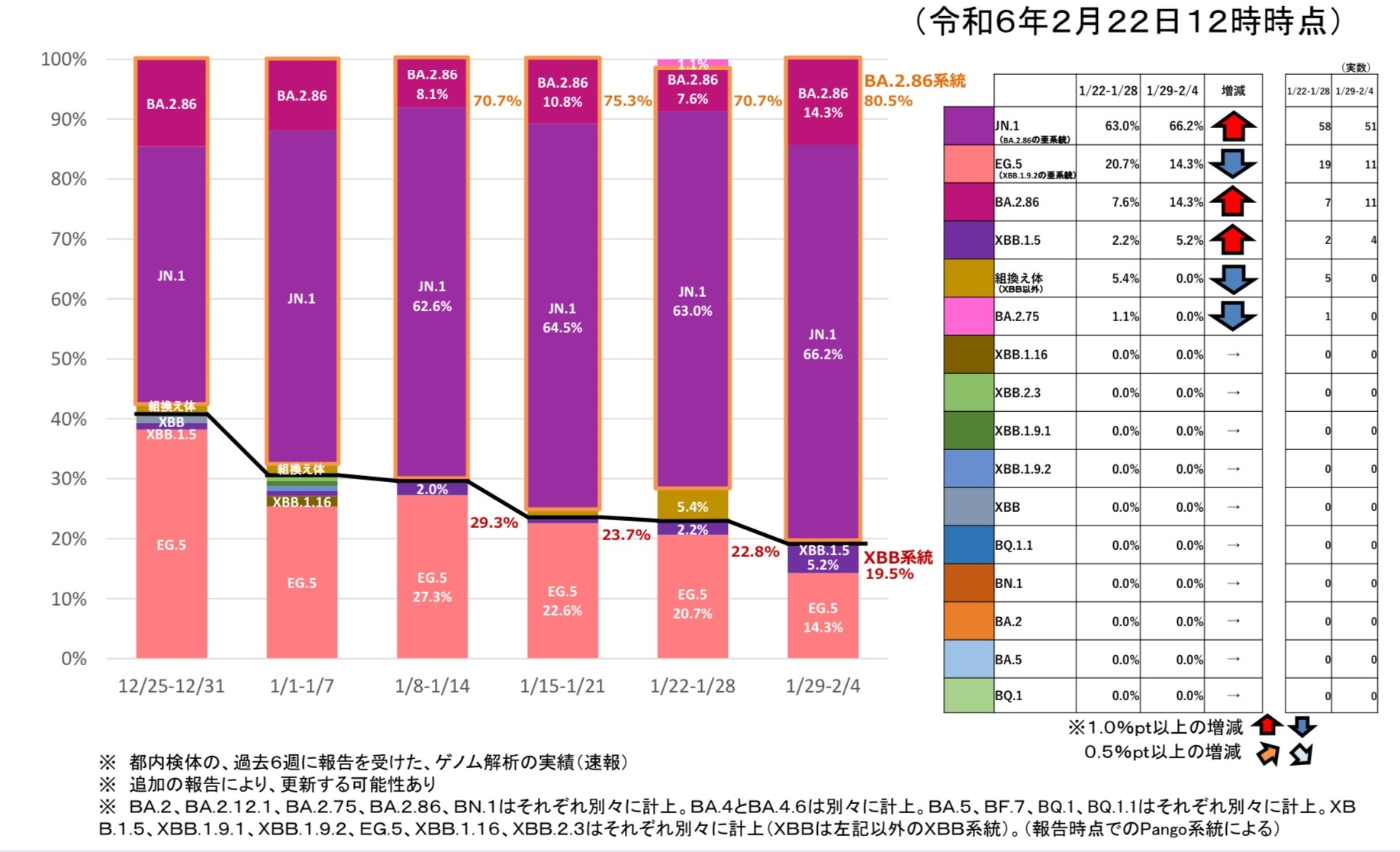
東京都の過去のモニタリング情報はコチラ:▶ ▶ ▶
オミクロン株の分岐状況:SARS-CoV-2: genome sequence prevalence and growth rate